
筛选支气管哮喘核心差异表达基因并对其进行生物信息学分析。
从基因表达数据库(GEO)下载哮喘患者巨噬细胞基因芯片数据GSE22528,该数据集包括了10份人肺泡灌洗液的转录组信息,其中哮喘患者和对照个体各5份。采用R 4.0.4软件筛选差异表达基因(DEGs)。利用DAVID 6.8数据库对筛选出的DEGs进行基因本体(GO)功能和京都基因与基因组百科全书(KEGG)通路富集分析。利用STRING在线数据库对DEGs编码蛋白构建蛋白互作网络(PPI),采用Cytoscape软件构建核心模块并确定核心DEGs。
肺泡灌洗液标本均来自加拿大白种人,哮喘患者和对照个体年龄范围分别为20~37和18~36岁,各组男性均为3例。哮喘患者上调基因449个,下调基因47个。GO分析显示:哮喘患者上调基因主要涉及对未折叠蛋白的反应等生物过程,分子功能集中于未折叠蛋白和生长因子的绑定;下调基因主要涉及组蛋白脱乙酰作用和泛素介导的蛋白质降解等生物过程,分子功能集中于组蛋白脱乙酰酶活性。KEGG通路富集分析显示:通路主要由上调基因富集,涉及Hippo信号通路、肥厚型心肌病、雌激素信号通路、致心律失常性右心室心肌病、基底细胞癌、神经活化的受体配体相互作用、扩张型心肌病和黏附连接等信号通路。PPI分析共得到两个核心模块,筛选出14个核心DEGs,分别为促黑色素聚集激素(PMCH)、孤啡肽前体(PNOC)、鞘氨醇-1-磷酸受体2(S1PR2)、鞘氨醇-1-磷酸受体5(S1PR5)、CC型趋化因子配体21(CCL21)、Kelch样蛋白25(KLHL25)、泛素结合酶E2V2(UBE2V2)、F-box蛋白17(FBXO17)、味觉受体2型成员3(TAS2R3)、生长抑素受体2(SSTR2)、代谢型谷氨酸受体2(GRM2)、李斯特E3泛素蛋白连接酶1(LTN1)、LIM域特有蛋白7(LMO7)和环指蛋白19A基因(RNF19A),其中LTN1和UBE2V2下调,其余均上调。
哮喘患者与对照个体存在DEGs、PMCH、PNOC、S1PR2、S1PR5和CCL21基因等可能为哮喘发病机制中的核心基因。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
支气管哮喘(简称哮喘)是一种以气道炎症、气道高反应性、可逆性气流阻塞和气道重塑为特征的常见慢性气道疾病[1, 2],其中肺泡巨噬细胞与哮喘关系密切,可通过多种方式引起气道炎症、损伤和重塑[3],影响哮喘患者的临床表型和病情[2]。本研究利用美国国家生物技术信息中心的基因表达数据库(GEO)(https://www.ncbi.nlm.nih.gov/geo/),筛选支气管哮喘差异表达基因(DEGs),为探讨支气管哮喘发病机制提供依据。
1.资料:源于美国国家生物技术信息中心GEO芯片平台的基因表达谱芯片(GSE22528)数据集,该数据集包括了10份人肺泡灌洗液的转录组信息,其中哮喘患者和无哮喘及过敏的对照个体各5份。
2.方法:(1)DEGs筛选:采用R 4.0.4软件对GSE22528数据集进行鲁棒多阵列分析,对数据进行背景校正、分位数归一化和中值抛光,筛选哮喘患者相对于对照个体的DEGs(基因上调或下调差异倍数>1.2且P<0.05);(2)基因本体(GO)功能注释和京都基因与基因组百科全书(KEGG)通路富集分析:利用DAVID 6.8数据库(https://david.ncifcrf.gov/)对筛选出的DEGs进行GO功能注释和KEGG通路富集分析;(3)蛋白互作网络(PPI)构建和核心DEGs筛选:利用STRING在线数据库(https://www.string-db.org/)对DEGs编码蛋白进行PPI构建,筛选联合评分>0.9的高置信度互作节点数据,将构建得到的PPI网络导入Cytoscape 3.7.2软件,进行网络数据集成、分析和可视化处理,采用分子复合物检测分析插件(MCODE)构建核心模块;采用cytoHubba插件分别以最大团中心性(MCC)、最大邻居连通度(MNC)和最大邻居连通密度(DMNC)3种算法选择得分前20位基因,在韦恩图中交集为核心DEGs。
3.统计学分析:DEGs利用t检验的P值和差异倍数进行筛选,筛选标准为上调或者下调差异倍数>1.2且P<0.05。对DEGs进行GO及KEGG分析,双侧检验,检验水准α=0.05。采用R 4.0.4软件对哮喘患者和对照个体的DEGs绘制热图。
1.基本情况:肺泡灌洗液标本均来自加拿大白种人,哮喘患者和对照个体年龄范围分别为20~37和18~36岁,其中男性均为3例(名)。哮喘患者上调和下调基因分别为449和47个。对哮喘患者和对照个体的上调及下调前40位DEGs绘制热图,红色代表上调,蓝色代表下调,显示两组人群中多数DEGs呈反向变化,说明哮喘患者相较于对照组个体表达基因的上调或下调趋势是具有明显差异的(图1)。
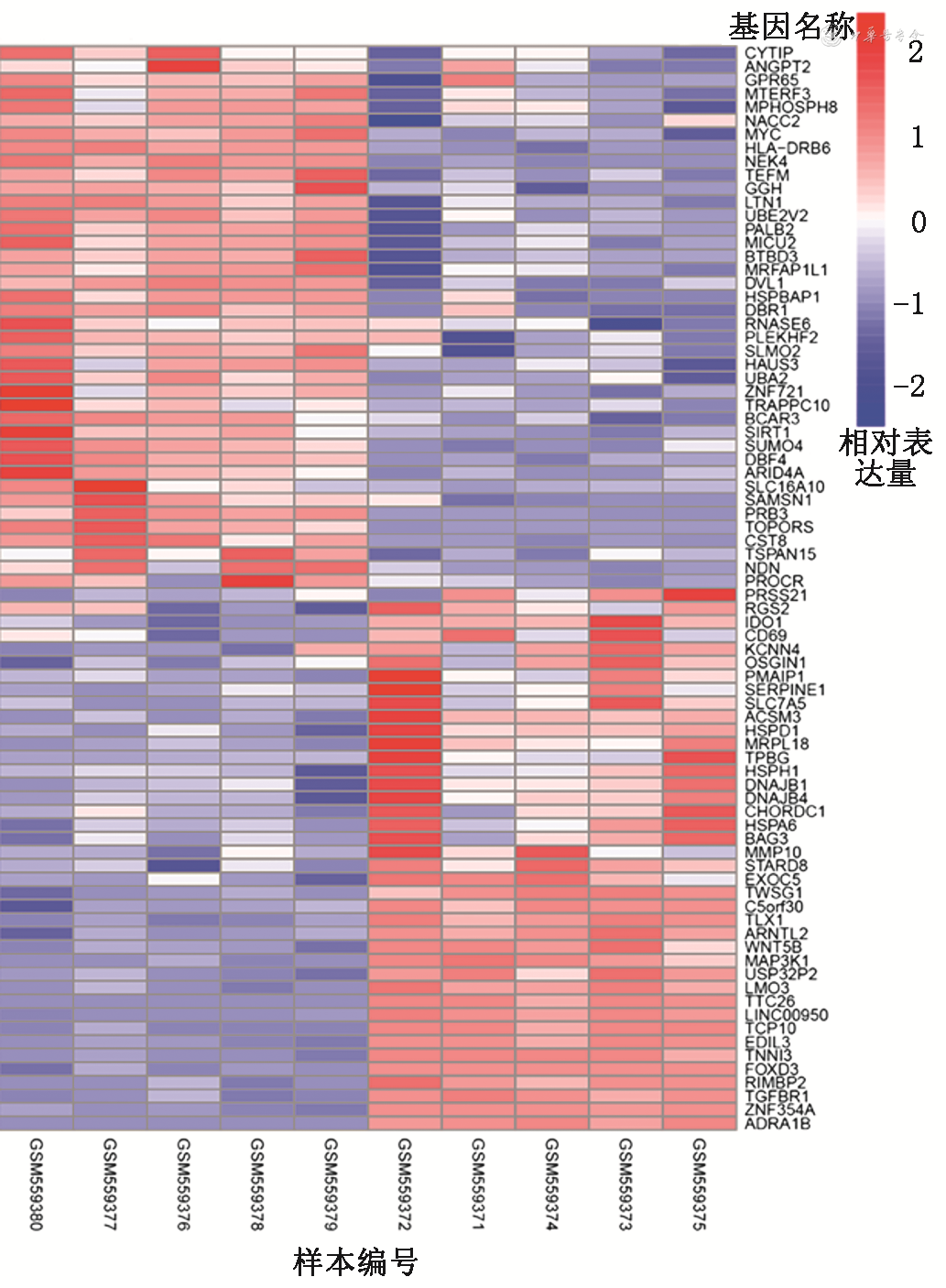

注:GSM559371~GSM559375为对照个体;GSM559376~GSM559380为哮喘患者
2.GO功能注释和KEGG通路富集分析:GO功能注释包括生物学过程、细胞组成和分子功能3部分。GO分析显示,哮喘患者上调基因主要涉及对未折叠蛋白的反应等生物过程,细胞组成包括细胞表面和质膜表面,分子功能集中于未折叠蛋白和生长因子绑定;哮喘患者下调基因主要涉及组蛋白脱乙酰作用和泛素介导的蛋白质降解等生物过程,细胞组成包括细胞核和核异染色质,分子功能集中于组蛋白脱乙酰酶活性(表1)。KEGG通路富集分析显示,通路主要由上调基因富集,涉及Hippo信号通路、肥厚型心肌病、雌激素信号通路、致心律失常性右心室心肌病、基底细胞癌、神经活化的受体配体相互作用、扩张型心肌病和黏附连接等信号通路。

哮喘患者与对照个体差异表达基因的基因本体(GO)功能富集分析
哮喘患者与对照个体差异表达基因的基因本体(GO)功能富集分析
| GO序号 | GO分类 | GO注释 | 调节类型 | 富集基因数 | P值 |
|---|---|---|---|---|---|
| GO:0006986 | 生物过程 | 对未折叠蛋白的反应 | 上调 | 11 | <0.001 |
| GO:0009986 | 细胞组成 | 细胞表面 | 上调 | 28 | <0.001 |
| GO:0051082 | 分子功能 | 未折叠蛋白的绑定 | 上调 | 11 | <0.001 |
| GO:0019838 | 分子功能 | 生长因子绑定 | 上调 | 6 | <0.001 |
| GO:0005886 | 细胞组成 | 质膜表面 | 上调 | 122 | <0.001 |
| GO:0004407 | 分子功能 | 组蛋白脱乙酰酶的活动 | 下调 | 3 | 0.004 |
| GO:0016575 | 生物过程 | 组蛋白脱乙酰作用 | 下调 | 3 | 0.006 |
| GO:0043161 | 生物过程 | 蛋白酶体介导的泛素依赖的蛋白质分解代谢过程 | 下调 | 4 | 0.013 |
| GO:0005634 | 细胞组成 | 细胞核 | 下调 | 19 | 0.038 |
| GO:0005720 | 细胞组成 | 核异染色质 | 下调 | 2 | 0.048 |
3.PPI构建和核心DEGs筛选:共构建两个可视化核心模块,各自包含8个DEGs,节点颜色按度值大小排列由浅至深,度值是指与周围节点相互作用的数目,度值越高,说明其与越多其他蛋白具有相关性,越可能是核心蛋白(图2)。通过MCC、MNC和DMNC 3种算法取交集获得14个核心DEGs,分别为促黑色素聚集激素(PMCH)、孤啡肽前体(PNOC)、鞘氨醇-1-磷酸受体2(S1PR2)、鞘氨醇-1-磷酸受体5(S1PR5)、CC型趋化因子配体21(CCL21)、Kelch样蛋白25(KLHL25)、泛素结合酶E2V2(UBE2V2)、F-box蛋白17(FBXO17)、味觉受体2型成员3(TAS2R3)、生长抑素受体2(SSTR2)、代谢型谷氨酸受体2(GRM2)、李斯特E3泛素蛋白连接酶1基因(LTN1)、LIM域特有蛋白7基因(LMO7)和环指蛋白19A基因(RNF19A)。
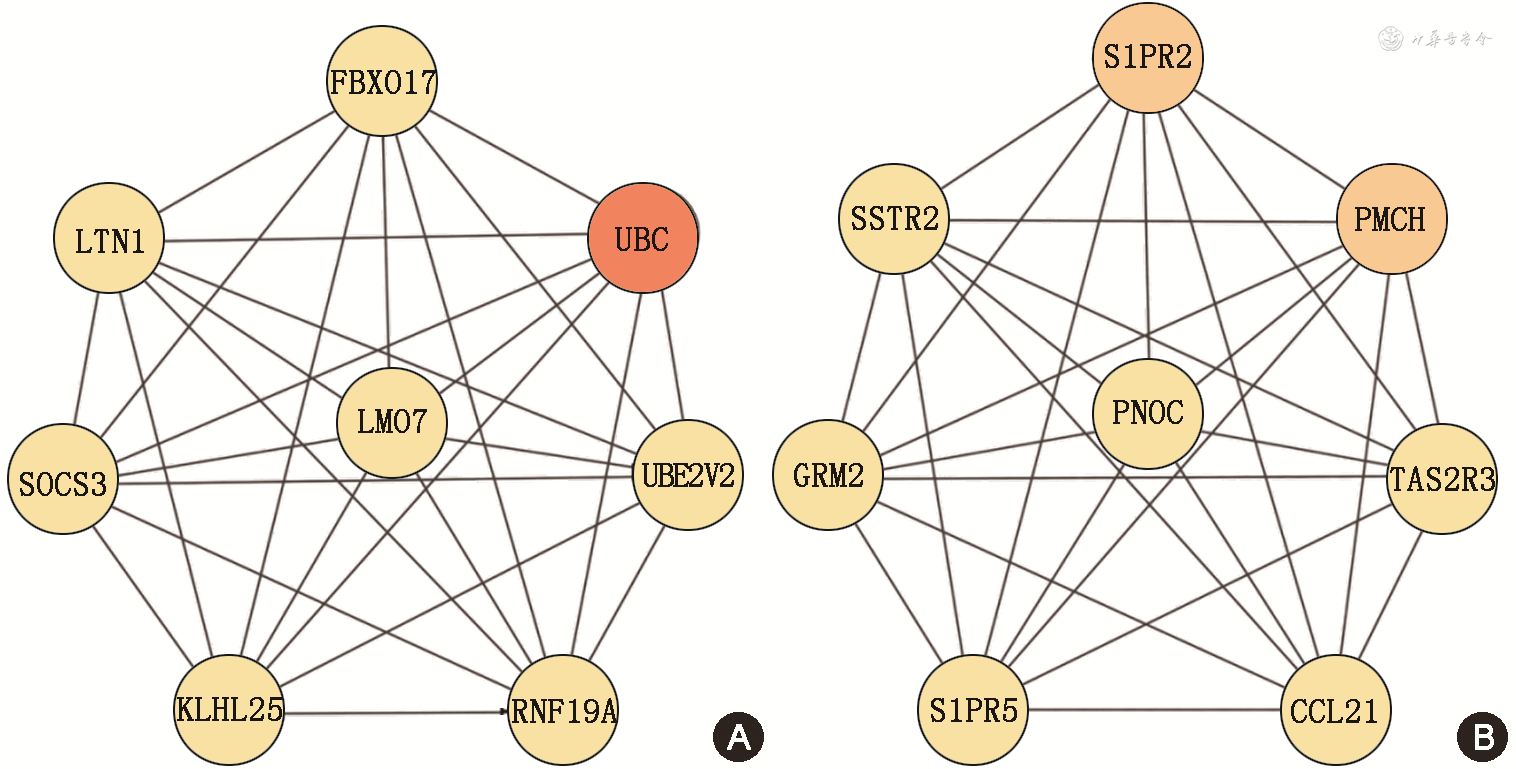

注:LTN1为李斯特E3泛素蛋白连接酶1;FBXO17为F-box蛋白17;UBC为泛素C;SOCS3为细胞因子信号转导抑制因子3;LMO7为LIM域特有蛋白7;KLHL25为Kelch样蛋白25;UBE2V2为泛素结合酶E2V2;RNF19A为环指蛋白19A;SSTR2为生长抑素受体2;S1PR2为鞘氨醇-1-磷酸受体2;PMCH为促黑色素聚集激素;PNOC为孤啡肽前体;GRM2为代谢型谷氨酸受体2;TAS2R3为味觉受体2型成员3;S1PR5为鞘氨醇-1-磷酸受体5;CCL21为CC型趋化因子配体21
基于基因数据库分析疾病相关DEGs已成为现代生物医学热点研究方法。本研究利用GEO芯片平台的基因表达谱芯片(GSE22528)数据集,获得哮喘患者14个核心DEGs,分别为PMCH、PNOC、S1PR2、S1PR5、CCL21、KLHL25、UBE2V2、FBXO17、TAS2R3、SSTR2、GRM2、LTN1、LMO7和RNF19A基因,其中LTN1和UBE2V2为下调,其他均为上调。
核心DEGs与哮喘的关系已有相应研究。其中,PMCH基因位于染色体12号q23.1区域,由TH2细胞选择性表达,调控体内过敏性炎症和能量代谢的联系,可能促进肥胖患者发生哮喘[4];PNOC蛋白受体在人类免疫细胞和气道细胞大量表达[5];Singh等[5]发现孤啡肽抑制气道高反应性、肺内嗜酸性细胞与肥大细胞迁移和炎症介质释放;肥大细胞和树突状细胞分别表达S1PR2和S1PR5基因亚型,S1PR蛋白则与S1P(鞘氨醇-1-磷酸)相结合,启动下游的生物过程[6];Liu等[7]发现S1P蛋白参与了哮喘和气道重塑,通过抑制S1P受体2调节气道平滑肌细胞自噬,缓解过敏性哮喘;S1P受体蛋白5对细胞外S1P蛋白做出反应,可导致细胞骨架的改变[8];一项荟萃分析结果表明,KLHL25基因存在于压力或暴力相关CpG岛中,该基因也与变应性哮喘有关[9]。
核心DEGs与肺泡巨噬细胞的关系亦有报道。活化的T淋巴细胞和内皮细胞所产生的CCL21蛋白作用于树突状细胞,可促进抗原呈递和巨噬细胞分化,与哮喘严重程度相关[10, 11],Xuan等[12]观察到CCL21蛋白对肺泡巨噬细胞M1/M2极化发挥重要作用,导致M1方向的极化;肺泡巨噬细胞可表达UBE2V2蛋白,介导靶基因转录激活[13];FBXO17蛋白调控肺泡巨噬细胞白细胞介素6(IL-6)的表达,阻断FBXO17蛋白表达可减少IL-6的产生,过表达则IL-6明显增加[14]。
然而,还有一些核心DEGs目前尚无其与哮喘的相关研究报道。LMO7蛋白在肺上皮细胞的顶端表面广泛表达,在上皮细胞的黏附连接形成中发挥作用[15],还与肺腺癌的发生和预后呈负相关[15, 16];UBE2V2基因是肺腺癌的致病基因且与预后相关[17];RNF19A蛋白定位于路易体,与神经元上的突触蛋白相互作用,与多种神经退行性疾病相关[18, 19];LTN1蛋白是核糖体蛋白质量控制的核心部分,其突变与神经变性相关[20];GRM2蛋白主要在神经元中表达,与中枢神经系统疾病相关[21];SSTR2蛋白与多种神经内分泌肿瘤相关,其与生长抑素结合,可发挥细胞抑制和细胞毒性作用[22];TAS2R3蛋白参与细胞膜上的信号转导,在苦味感知中发挥功能[23]。这些核心DEGs与哮喘的关系尚需进一步研究。
随着对哮喘发病机制研究的深入,相应的生物靶向制剂也得到了更好的发展与使用[24]。本研究探讨了巨噬细胞方向基因层面的差异,可能为支气管哮喘巨噬细胞方面未来的靶向治疗提供一定的科研依据和理论基础,但局限性在于还缺乏相关生物学实验证实,下一步可能需要在相关的细胞及动物模型中对上核心基因进行功能验证。
所有作者均声明不存在利益冲突



























