
传统以检测特定生化标志物为技术手段的新生儿筛查(newborn screening,NBS)作为社会健康第三级防控体系,在出生缺陷防控领域发挥了很好的作用,但筛查病种较少,部分病种存在一定程度的假阳性和假阴性。而遗传检测技术的快速发展以及在遗传性疾病诊断中经验的积累,为遗传病NBS提供了新的技术选择。将遗传检测技术引入NBS已成为大势所趋。国内外学者在新生儿基因筛查领域进行了积极的尝试并积累了一些经验和教训。为尽可能最大程度保护基因筛查新生儿及其家庭权益,在总结国内外前期探索性基因筛查经验的基础上,业内专家共同探讨拟定基于高通量测序技术(next generation sequencing,NGS)的中国单基因病NBS(简称新生儿基因筛查)共识,以规范新生儿基因筛查体系,指导NGS在新生儿基因筛查领域的应用。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
新生儿筛查(newborn screening,NBS)是指在新生儿期对严重危害新生儿健康的先天性、遗传性疾病施行专项检查,提供早期诊断和治疗的母婴保健技术[1]。NBS发展至今已有60余年的历史,技术的进步极大地推动了NBS的发展,无论从筛查病种、筛查效率都获得很大程度提升[2,3,4,5,6,7]。几十年实践经验证实NBS通过筛查早期诊治很大程度上改善了患者的预后,改善了患者家庭的生活质量,获得很好的家庭、社会效益[2,3,4,5,6,7],但仍存在以下尚待解决的问题:(1)筛查病种较少,还有很多可防、可治的遗传性疾病未纳入筛查体系;(2)部分病种筛查效率较低。虽经各国研究者的多方面尝试,筛查阳性预测值获得较大改善[8,9,10],但初筛生化标志物正常范围案例的假阴性问题仍未解决。近年各种遗传检测技术,尤其高通量测序技术(next generation sequencing,NGS)的飞速发展,无论在测序费用的减低和遗传病诊断经验的积累等方面均取得很大突破;同时,随着国内外相关专家共识及指南的推行,NGS技术在检测准确性、报告速度、报告规范性等方面亦获得极大提升[11,12,13,14,15,16,17,18,19,20,21,22,23]。国内外学者在将遗传检测技术引入NBS或高危患儿筛查领域均进行了探索性研究并积累了一定程度的经验和教训,为新生儿基因筛查提供了循证依据[24,25,26,27,28,29,30,31,32,33,34]。与传统NBS比较,基因筛查在目前尚无可靠生化标志物的病种上有明显优势;尤其NGS具有更高通量,有利于扩大筛查病种及缩短疾病确诊时间;而早期获得先证者基因型,有利于精准诊治及遗传疾病防控前移[24,25,26,27,28,29,30,31,32,33,34]。但研究也揭示了基因筛查存在的问题:(1)对致病基因意义未明位点解读困难;(2)基于目前对人类遗传病的认知程度,基因组测序技术用于新生儿群体筛查的优势尚未明确;(3)社会伦理问题等[25,26,27,28,29,30,31,32,33,34]。目前国内针对新生儿群体的探索性基因筛查项目日益增多,为最大程度保障筛查新生儿及其家庭的权益,业内专家本着科学性、前瞻性、技术保障、伦理优先的准则,共同探讨建立NGS在单基因病NBS(简称新生儿基因筛查)应用中的专家共识,希望通过本共识进一步规范新生儿基因筛查体系,指导NGS技术在新生儿基因筛查领域的应用。
新生儿基因筛查场地、人员、设备、试剂及管理流程均需符合目前新生儿疾病筛查相关的各项规定[1,35]。同时该机构需符合《医疗机构临床基因扩增检验实验室管理办法(2010年版)》及《医疗机构临床基因扩增检验工作导则(2010年版)》要求,或有稳定的具备上述要求的合作机构[36]。
在符合NBS相关技术规范对人员要求的基础上,实验室负责人需符合《医疗机构临床基因扩增检验实验室管理办法》要求并具有临床基因扩增实验室管理经验[36]。所有人员需严格按照相关规定要求的职责、诊治技术规程操作。
NBS是集组织管理、实验室技术、临床诊断和公众教育等为一体的社会健康项目,涉及多学科,要有一套完善的数据分析及信息管理系统。筛查机构需建立完善的转诊机制,以满足可疑阳性和阳性患儿的诊治、随访需求。合作机构应当与NBS机构签订协议明确各自的责任和义务,落实后续检测与诊断服务。相关医疗机构要严格按照医学伦理原则,自觉维护新生儿权益,保护个人及家庭隐私。
(1)疾病危害严重,可导致残疾或死亡;(2)疾病的发生率相对较高,且发病机制与异常产物已阐明;(3)疾病早期无特殊症状,但有实验室指标能显示阳性;(4)有准确可靠、适合在新生儿群体中大规模进行筛查的方法,假阳性率和假阴性率均较低,并易为家长所接受;(5)已建立有效治疗方法,特别是通过早期治疗,能逆转或减慢疾病发展,或者改善其预后;(6)筛查费用、医学治疗效果及社会经济效益的比例合理;(7)虽目前尚无有效治疗方法,但符合上述除第5条外其他原则,且对家庭再生育指导有帮助,致病基因明确的单基因疾病。
在符合上述标准的基础上,充分利用遗传检测技术高通量的优势,结合本地遗传病患病率/发病率,选择致病基因明确、针对新生儿期可诊断并在儿童期有有效治疗或干预[或符合病种选择原则(7)]的单基因遗传病进行筛查,如G6PD、DUOX2、GJB2、PAH、ETFDH、MMACHC、SLC25A13、GCDH等基因变异已证实为中国人群较常见基因[37,38,39]。建议保留目前常规NBS病种相关基因。纳入的筛查基因原则上需符合临床基因组资源中心(Clinical Genome Resource,ClinGen)的确定级标准(definitive cause gene)[40]。建议尽可能多的涵盖临床常见遗传性单基因病,同时需兼顾基因检测技术的灵敏度,整个筛查项目的成本/效益比。
基于上述两点原则,综合现有我国和国际成熟及探索性新生儿单基因疾病筛查经验,优先推荐病种包括遗传代谢病及各系统15类66种单基因遗传病(附录1)。实际操作过程中可根据本地具体情况酌情增减,或制定包含不同基因数量的靶向测序包供家长知情同意情况下结合自身需求自主选择。
如果新生儿同时进行了传统生化/串联质谱筛查和基因筛查,并且两种筛查的目标疾病有交叉,结合二者的结果可以更加有效和准确地检测到可能患病的新生儿。生化/串联质谱筛查的结果可以作为基因筛查结果解读的辅助证据,而基因筛查的结果也可以帮助排查可能的生化/串联质谱筛查的假阳性或者假阴性结果。为了两种筛查结果有效联合分析,传统NBS实验室与新生儿基因筛查实验室需要建立标准的联络机制,保证结果和数据的及时准确传输。
医务人员要严格按照医学伦理学原则,充分告知新生儿监护人目标筛查疾病、意义、检测项目的预期结果(如阳性、阴性、不确定结果)、费用、操作流程、检测风险等问题;告知NBS方法学的局限性,若有多种筛查方式,告知不同方式的优势和利弊(如基于微阵列芯片的技术方案,通常只针对预先筛选的较高频致病位点进行检测分析,对于其他的位点则无法检出。基于多重PCR结合NGS的技术方案,除了能覆盖预先筛选的位点,根据设计的扩增子的大小也能覆盖这些位点附近的部分区域。而基于液相捕获结合二代测序的方法可以实现对绝大部分基因蛋白编码区域进行分析,但对非编码区域通常不能覆盖,因此存在不能检出位于这些区域的致病位点的可能性。对于高度重复序列或同源序列区域、大片段的缺失或插入变异以及特殊的复杂变异存在假阴性和假阳性可能);告知NBS筛查、随访流程。告知筛查结果可能对新生儿及亲属的影响;告知相关法律权限。在充分告知新生儿监护人并知情同意情况下,书面签署知情同意书。签署知情同意书后方能进入标本采集、检测流程。知情同意书应一式两份,一份筛查机构留存,一份监护人留存。
新生儿基本资料信息的采集,样本的采集及转运,样本采集注意事项等参照《新生儿疾病筛查技术规范(2010年版)》[1]以及《新生儿疾病筛查滤纸血片采集和递送及保存专家共识》[41]执行,建议与常规已开展NBS标本同时采取。注意采集样本量需满足基因筛查项目所需。
NGS的特点:(1)大规模平行测序,通量高,分析灵敏度和特异性较高;(2)有定量功能,即样品中某一基因序列被测序的次数反映了样品中这个基因的丰度,因此可进行基因组拷贝数分析;(3)相比一代测序技术,其单碱基测序成本明显下降。NGS技术对同时涉及多个基因、多种变异类型以及罕见变异的出生缺陷和遗传性疾病的分子诊断具有非常明显的优势,在鉴定疾病新致病基因方面也非常有效[42,43]。NGS技术按其检测范围大小不同,可分为疾病靶向基因包测序、全外显子组测序(WES)以及全基因组测序(WGS)等。综合考虑NBS病种纳入标准及成本/效益比,建议现阶段采用疾病靶向基因包。
实验室在收到样本后应当尽快完成DNA抽提并对DNA质量进行质检。可以采用吸光度法检测DNA的纯度(A260/A280值1.6~2.2),并通过琼脂糖凝胶电泳检查DNA的完整性(主带明显,无明显降解,无明显的蛋白质、多糖和RNA污染)。DNA样本可以短期保存在4 ℃环境,长期保存环境温度不高于-20 ℃,同时避免不必要的反复冻融。
目标基因文库构建是整个NGS实验过程中重要的一环,其质量的好坏将直接影响后续的测序数据质量。单个8 mm血斑DNA产量通常不超过200 ng,因此适宜的建库方法是通过多重PCR扩增捕获目标序列。如果可从多个血斑中提取足够的DNA样本,则可通过目前较为常用的液相杂交捕获方法建库。对构建好的文库需检测浓度或总量、片段长度及分布等,具体标准需根据检测策略的不同而制定。
目前市场上有不同通量、读长及研究适配的NGS仪器,测序时应根据检测的样本量和质量要求,选择适宜的测序平台与方案,以保证测序数据能够满足数据质量及靶向区域覆盖度等需求。测序数据单碱基质量评估指数Q30需达到一定标准,并根据不同检测策略对基因变异判定所需的数据量制定所需的平均测序深度[44,45]。
NGS生物信息学分析流程主要包括序列比对、变异检测、变异注释、变异筛选过滤以及相关质控统计等步骤。各实验室需建立测序质量标准,评价指标包括目标区域的覆盖度、平均深度、重复序列比例、比对到目标区域的读句比例、目标区域每个碱基的测序深度等。基因变异分析环节涉及变异初筛、表型匹配和变异致病性判读等多个步骤,各实验室需根据行业相关指南编写数据解读标准操作流程(standard operating procedures,SOPs)[36],要求相关人员在充分的培训和考核后方可进行数据解读和报告出具。
实验室对获得的疑似阳性检测结果,如对单核苷酸变异(SNV),通常需要进行Sanger测序验证。如果实验室建立了成熟的实验方法和质控体系,依据既往数据,对于能够长期稳定检出的位点可以不进行验证,这样有助于缩短报告时间和检测成本。但对于一些比较复杂的变异类型,如小插入和缺失(indel),则需要进行验证。建议实验室在没有获得充分检测经验和完善质控体系之前,对所有获得的疑似阳性结果进行验证。
各种分子诊断技术特别是NGS在遗传病诊断中的广泛应用,发现了大量的可能与遗传病相关的变异。如何准确的评估和分析这些变异的临床意义,成为了分子诊断领域的巨大挑战,而这些挑战推动了基因变异致病性分析和判读的标准化。2015年,美国医学遗传学会(American College of Medical Genetics,ACMG)联合分子病理学学会(Association of Molecular Pathology,AMP),根据对多个临床分子诊断实验室的调查以及专家的建议,推出了基因序列变异的致病性解读指南[46]。此后,ClinGen专家组在此指南的基础上,继续进行了补充和优化,发表了一系列建议[40]。这些指南、建议和共识,被很多临床分子诊断实验室应用,极大地促进了基因变异致病性判读的标准化,提升了位点解读结果的一致性。对于新生儿基因筛查检测到的基因变异,建议各实验室参考已有的位点判读指南、共识和技术标准,建立检测结果解读的规范标准流程,保证解读的一致性和准确性。
与通常的遗传病诊断检测服务不同,新生儿基因筛查的目标人群主要是表型正常的新生儿,因此无法根据受检者的临床表型对检测到的位点进行评估。因此,已报道的证据(主要是已发表文献和公共数据库)、实验室内部的位点数据库以及生物信息学的预测结果,是对这些位点进行判读的主要证据。对这些证据的充分挖掘和完整分析,是对位点进行充分准确解读的关键。此外,一些商业数据库也能提供更多信息[47,48,49]。
新生儿基因筛查报告的基因和位点,应当遵循为受检新生儿和家庭利益最大化的原则,需要综合考虑报告内容的临床价值和临床咨询的可行性,也要避免不必要的给受检家庭带来焦虑和伦理问题。因此,对于新生儿基因筛查报告的范围,建议如下:(1)谨慎选择报告的基因,需要有充分的证据表明该基因会导致高外显率的儿童期疾病(<18岁发病);(2)可以考虑报告在儿童期进行干预可能避免后续重大疾病发生的相关中等证据或中等外显率的基因[48];(3)建议仅报告致病和疑似致病的变异,避免报告内容的不确定性,减少临床咨询的难度;(4)如果在常染色体隐性遗传基因病种只发现1个致病或可能致病位点,报告可能为携带,但需结合其他临床指征或家族史,若符合该基因相关表型,则建议进行诊断性的检测;(5)如果位点导致较轻的症状或发病较晚(>18岁),或有文献支持位点存在外显不全的情况,则建议不报告,或者结合家族史和其他临床指征综合考虑后报告;(6)X连锁基因致病位点的女性杂合携带者,可能存在X染色体失活偏好而导致非经典表型,但密切跟踪和随访可以显著降低发病风险,因此建议报告这类变异,但需要经遗传专科医师进行充分的遗传咨询。
新生儿基因筛查报告的格式,与遗传病检测报告格式类似,建议包括如下信息:(1)基本信息:受检者唯一识别码、出生日期、性别、父母信息、样本类型、采样日期、检测日期、报告日期、送检医师和医院信息、检测实验室信息;(2)检测结果:标准基因名、参考基因组坐标、转录本编号、核苷酸改变、氨基酸改变、杂合性、致病性、相关疾病和信息介绍、遗传模式;(3)其他信息:临床和遗传咨询建议、技术质控参数、检测覆盖的基因或者变异位点、临床和技术局限性和参考文献等。
根据上述新生儿基因筛查报告原则判读为阳性的新生儿基因筛查案例以及基因及传统生化联合筛查项目中新生儿基因筛查阴性但合并传统生化筛查可疑阳性案例均需召回。筛查机构负责阳性/可疑阳性案例的召回工作。
上述案例需转诊至具遗传学背景的咨询师、专科医师处进一步做变异位点父母验证及其他辅助性检查以协助诊断;确诊患者需要包括多学科医务人员、社会服务人员、慈善机构的共同参与,做好治疗、遗传咨询及随访工作,以确保患者及家庭从NBS中获益。
(1)在遗传咨询过程中必须遵循公正、尊重自主、有益、无伤害的伦理原则。新生儿基因筛查的对象是新生儿,应充分告知监护人NBS的益处及筛查的局限性,引导其在充分知情的情况下代表孩子做出有利的选择。充分告知可能因为假阳性带来的心理影响和假阴性对婴儿的伤害。(2)遗传咨询过程中必须保证相关信息的安全,避免可能会对筛查新生儿上学、就业和婚姻等带来不利影响。(3)筛查前充分沟通遗传信息的归属、是否用于科学研究等可能涉及的问题。
(童凡 王剑 肖锐 执笔)
参与本共识制定、讨论专家(按姓氏拼音排序,排名不分先后):顾学范(上海交通大学医学院附属新华医院);韩连书(上海交通大学医学院附属新华医院);黑明燕(首都医科大学附属北京儿童医院);黄新文(浙江大学医学院附属儿童医院);黄永兰(广州妇女儿童医学中心);童凡(浙江大学医学院附属儿童医院);吴冰冰(复旦大学附属儿科医院);吴鼎文(浙江大学医学院附属儿童医院);王华(湖南省儿童医院);王剑(上海交通大学医学院附属国际和平妇幼保健院);文伟(深圳市妇幼保健院);许争峰(南京医科大学附属妇女医院,南京市妇幼保健院);肖锐(出生缺陷防控关键技术国家工程实验室);杨琳(复旦大学附属儿科医院);杨茹莱(浙江大学医学院附属儿童医院);袁天明(浙江大学医学院附属儿童医院);余永国(上海交通大学医学院附属新华医院);杨艳玲(北京大学第一医院);邹朝春(浙江大学医学院附属儿童医院);邹卉(山东第一医学院附属妇幼保健院,济南市妇幼保健院);邹琳(上海市儿童医院);周文浩(复旦大学附属儿科医院);张拥军(上海交通大学医学院附属新华医院);赵正言(浙江大学医学院附属儿童医院)
所有作者均声明不存在利益冲突
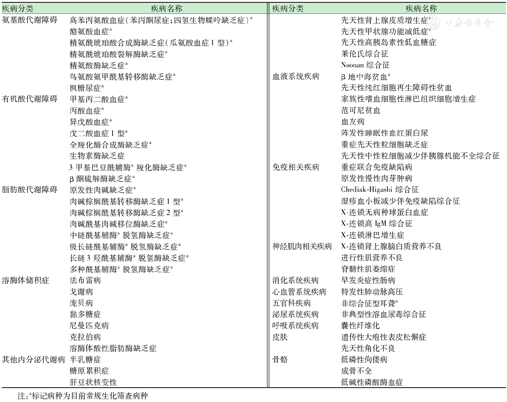
| 疾病分类 | 疾病名称 |
|---|---|
| 氨基酸代谢障碍 | 高苯丙氨酸血症(苯丙酮尿症;四氢生物蝶呤缺乏症)a |
| 酪氨酸血症a | |
| 精氨酰琥珀酸合成酶缺乏症(瓜氨酸血症1型)a | |
| 精氨酰琥珀酸裂解酶缺乏症a | |
| 精氨酸酶缺乏症a | |
| 鸟氨酸氨甲酰基转移酶缺乏症a | |
| 枫糖尿症a | |
| 有机酸代谢障碍 | 甲基丙二酸血症a |
| 丙酸血症a | |
| 异戊酸血症a | |
| 戊二酸血症1型a | |
| 全羧化酶合成酶缺乏症a | |
| 生物素酶缺乏症 | |
| 3甲基巴豆酰辅酶a羧化酶缺乏症a | |
| β酮硫解酶缺乏症a | |
| 脂肪酸代谢障碍 | 原发性肉碱缺乏症a |
| 肉碱棕榈酰基转移酶缺乏症1型a | |
| 肉碱棕榈酰基转移酶缺乏症2型a | |
| 肉碱酰基肉碱移位酶缺乏症a | |
| 中链酰基辅酶a脱氢酶缺乏症a | |
| 极长链酰基辅酶a脱氢酶缺乏症a | |
| 长链3羟酰基辅酶a脱氢酶缺乏症a | |
| 多种酰基辅酶a脱氢酶缺乏症a | |
| 溶酶体储积症 | 法布雷病 |
| 戈谢病 | |
| 庞贝病 | |
| 黏多糖症 | |
| 尼曼匹克病 | |
| 克拉伯病 | |
| 溶酶体酸性脂肪酶缺乏症 | |
| 其他内分泌代谢病 | 半乳糖症 |
| 糖原累积症 | |
| 肝豆状核变性 | |
| 先天性肾上腺皮质增生症a | |
| 先天性甲状腺功能减低症a | |
| 先天性高胰岛素性低血糖症 | |
| 莱伦氏综合征 | |
| Noonan综合征 | |
| 血液系统疾病 | β地中海贫血a |
| 先天性纯红细胞再生障碍性贫血 | |
| 家族性嗜血细胞性淋巴组织细胞增生症 | |
| 范可尼贫血 | |
| 血友病 | |
| 阵发性睡眠性血红蛋白尿 | |
| 重症先天性粒细胞缺乏症 | |
| 先天性中性粒细胞减少伴胰腺机能不全综合征 | |
| 免疫相关疾病 | 重症联合免疫缺陷病 |
| 原发性慢性肉芽肿病 | |
| Chediak-Higashi综合征 | |
| 湿疹血小板减少伴免疫缺陷综合征 | |
| X-连锁无病种球蛋白血症 | |
| X-连锁高IgM综合征 | |
| X-连锁淋巴增生症 | |
| 神经肌肉相关疾病 | X-连锁肾上腺脑白质营养不良 |
| 进行性肌营养不良 | |
| 脊髓性肌萎缩症 | |
| 消化系统疾病 | 早发炎症性肠病 |
| 心血管系统疾病 | 特发性肺动脉高压 |
| 五官科疾病 | 非综合征型耳聋a |
| 泌尿系统疾病 | 非典型性溶血尿毒综合征 |
| 呼吸系统疾病 | 囊性纤维化 |
| 皮肤 | 遗传性大疱性表皮松懈症 |
| 先天性角化不良 | |
| 骨骼 | 低磷性佝偻病 |
| 成骨不全 | |
| 低碱性磷酸酶血症 |
注:a标记病种为目前常规生化筛查病种



























第36页附录1右栏第18行“X-连锁无病种球蛋白血症”更正为“X-连锁无丙种球蛋白血症”。更正的同时表示歉意!