版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
近年来,表皮生长因子受体–酪氨酸激酶抑制剂(epidermal growth factor receptor–tyrosine kinase inhibitors, EGFR–TKIs)等靶向药物已经成为晚期非小细胞肺癌(NSCLC)重要的治疗方式之一。在EGFR基因突变的晚期NSCLC患者,系列国际国内多中心临床研究已证实EGFR靶向治疗能显著降低疾病进展或死亡风险、改善患者生活质量。而在EGFR基因未发生突变的肺癌患者中,临床研究证据提示不宜使用靶向药物。这些临床研究和系统分析均指出EGFR基因突变检测是晚期肺癌患者使用EGFR–TKIs治疗的先决条件。
鉴于多种原因,临床实践中EGFR基因突变的肺癌组织受检率并不高,导致基于驱动基因变异靶点的个体化治疗策略难以实施。多项回顾性和前瞻性研究表明,当肿瘤组织难以获取时,血液循环游离或肿瘤DNA(cf/ctDNA)检测是突变分析合适的替代选择。
中国国家食品药品监督管理总局(CFDA)在2015年2月已批准吉非替尼说明书更新,在推荐所有NSCLC患者的肿瘤组织都应进行EGFR基因突变检测基础上,如果肿瘤标本不可评估,则可使用从血液(血浆)标本中获得的ctDNA进行评估。
目前我国尚无规范化的血液EGFR基因突变基因检测的标准或指南。因此,为了规范中国肺癌患者血液EGFR基因突变的检测,特组织全国肺癌和病理学领域的知名专家共同制定本专家共识。
世界卫生组织下属的国际癌症研究机构(IARC)于2013年12月12日公布了2012年全球癌症流行病学最新数据(GLOBOCAN 2012):肺癌是世界范围内发病率(新发肺癌患者182万,占所有新发肿瘤病例的13%)和死亡率(死亡肺癌患者160万,占所有死亡肿瘤病例的19.4%)最高的恶性肿瘤[1]。在中国,肺癌的发病率和死亡率均位居所有恶性肿瘤的首位。2012年中国新发肺癌患者约65.3万,死亡患者约59.7万[2]。根据肺癌生物学特征,NSCLC占所有肺癌的80%~85%,肺腺癌的发病率已超过肺鳞癌[3]。
EGFR是一种跨膜酪氨酸激酶受体,该受体激酶域的激活对癌细胞增殖、生长的相关信号传递具有重要意义。EGFR基因两种主要的突变为19外显子缺失(约占45%)和21外显子L858R突变(约占40%)[3,4],二者均可导致酪氨酸激酶结构域活化,且均为EGFR–TKIs的敏感性突变。肺腺癌患者EGFR基因敏感突变阳性率在高加索人群约为10%,在亚裔人群和我国均为50%左右[5]。
在过去十多年间,EGFR–TKIs已逐渐成为EGFR基因突变的晚期NSCLC患者的一线标准治疗。具有里程碑意义的随机、对照、大样本Ⅲ期IPASS临床试验首次显示吉非替尼一线治疗EGFR基因突变晚期NSCLC患者的无进展生存时间、肿瘤缓解率和生活质量均优于标准化疗,并且具有良好的安全性[5]。随后,基于EGFR基因突变阳性患者进行的前瞻性Ⅲ期临床试验(NEJ002、WJOG3405、OPTIMAL、EURTAC及LUX–Lung3 /LUX–Lung6),通过与标准化疗比较,充分验证了EGFR基因突变是一线EGFR–TKIs治疗的重要靶点和关键性预测标志物[3,5]。另一方面,CTONG0806等临床研究和荟萃分析显示,在EGFR基因未发生突变的肺癌患者,因不能降低疾病进展或死亡风险,不宜使用EGFR靶向治疗[6,7]。因此,以组织和细胞学标本进行EGFR基因突变检测用于指导NSCLC患者EGFR–TKIs治疗已在全球形成共识,成为标准的临床实践[4]。
美国国立综合癌症网络(NCCN)指南建议:腺癌、大细胞癌及组织学类型无法确定等NSCLC应进行EGFR基因突变检测;非吸烟、小活检标本或混合型组织学类型的鳞癌患者也可考虑进行EGFR基因突变检测;可采用多种方法进行检测[3]。
欧洲临床肿瘤协会(ESMO)指南建议:晚期非鳞癌的NSCLC患者应进行EGFR突变检测;除了从不吸烟或者很少吸烟(每年<15包)的鳞癌患者外,已确诊鳞癌患者不推荐进行EGFR基因突变检测[8]。
美国临床肿瘤学会(ASCO)于2014年签署了一项美国病理学家联合学会(CAP)/国际肺癌研究协会(IASLC)/分子病理学协会(AMP)关于肺癌患者选择治疗靶点分子检测的临床实践指南,该指南明确指出:所有肺腺癌或者混合腺癌成分的肺癌患者,不考虑性别、种族和吸烟情况的特征,均应进行EGFR基因突变检测[9]。
我国NSCLC患者EGFR基因突变检测专家共识指出:所有NSCLC患者,不限组织学类型,只要条件许可,均应尝试进行基于肿瘤组织的EGFR基因突变检测[10]。
来自肿瘤部位的手术组织、活检组织以及细胞学标本,新鲜或甲醛固定石蜡包埋等处理的组织或细胞学标本,均可用于EGFR基因突变检测。临床取材方法主要包括纤维支气管镜下活检、经皮肺穿刺活检、胸水、手术、胸腔镜、淋巴结穿刺活检、支气管内超声引导细针穿刺活检(EBUS FNA)等[3,8,9,10]。
2015年欧洲肺癌大会(ELCC)上公布了一项全球10个国家562位肿瘤专家参与的关于晚期NSCLC患者EGFR基因突变检测和治疗策略选择的调查研究结果:19%的晚期NSCLC患者未进行EGFR基因突变检测,即使进行检测,仍有26%的欧洲患者在结果出来前就开始接受治疗[11]。未进行检测最常见的主要原因是病理组织学类型的限制(如鳞癌);其次是缺乏足够的肿瘤组织。检测周期过长亦是未行EGFR基因突变的另一原因。在部分检测中心,检测周期甚至可达数周[11]。此外,部分患者肿瘤内EGFR基因突变的异质性使基于基因检测的靶向治疗决策变得更加困难[12]。
我国的EGFR基因突变检测在肺癌患者中的受检率相对偏低,Xue等[13]报道2012年我国肺癌EGFR基因突变受检率为20%~30%,明显低于发达国家或地区的水平[14]。如何在我国提高EGFR等基因变异受检率是提升我国肺癌精准诊治的关键因素之一。我国的肺癌EGFR基因突变受检率低也是与临床组织获取受限、经济原因、医师与患者的精准诊治意识、药物昂贵及获得不易等多因素相关。
针对临床实践中肿瘤组织检测的种种局限,探寻作为其补充或替代的其他生物标本进行EGFR基因突变检测具有重要临床意义,有助于提高临床患者的总体EGFR基因受检率。
研究显示,大部分(并非全部)晚期NSCLC患者的血液中存在循环游离DNA(cfDNA, cell free DNA)[15,16,17]。cfDNA主要来源于凋亡或坏死的细胞,包括正常细胞和肿瘤细胞,如果来自肿瘤细胞称为循环肿瘤DNA(ctDNA)。但血液游离DNA片段通常较短,在晚期癌症患者血液中浓度极低,平均约为17 μg/L[18,19]。
既往系列研究已经在NSCLC患者的血浆或血清样本ctDNA中发现EGFR基因突变,初步显示外周血ctDNA EGFR基因突变检测及对EGFR–TKIs疗效预测的可行性[16,20,21]。IPASS研究通过扩增阻滞突变系统(amplification refractory mutation system, ARMS)方法检测日本亚组人群血清游离DNA的EGFR基因突变状况与临床疗效的研究数据显示,血清游离DNA EGFR基因突变阳性患者接受吉非替尼治疗较标准化疗患者疾病无进展生存期(PFS)显著延长[15]。IFUM研究也提供了在高加索人群中基于血浆游离DNA检测(ARMS检测)发现的EGFR基因突变阳性患者接受吉非替尼治疗NSCLC的疗效证据,在肿瘤组织阳性(此组包括血浆阳性与阴性患者)、肿瘤组织和血浆标本均阳性及肿瘤组织阳性而血浆标本阴性三组中,其客观缓解率分别为70%、76.9%和59.5%[22]。在FASTACT–Ⅱ研究中,血浆EGFR基因突变患者接受厄洛替尼与化疗联合治疗的客观缓解率(74.6%与19.7%)和无进展生存期均优于单独化疗组[23]。而230例中国晚期NSCLC组织与血浆游离DNA配对分析等研究同样显示血液标本EGFR基因突变对于预测靶向药物治疗的临床获益与组织突变者高度一致[20]。
与肿瘤组织相比,ctDNA中EGFR基因突变检测具有高度特异性(IPASS、IFUM和IGNITE研究中的特异度分别为100%、99.8%和97.2%),但敏感度相对较低(分别为43.1%、65.7%和49.6%);这可能与肿瘤分期、血液标本的处理、检测方法差异等相关[12,15,22,24]。这些特定技术下的血液EGFR基因突变检测的低敏感度提示在临床实践中应高度关注单一血液检测结果的假阴性情况。
欧洲药品管理局2014年9月已批准当难以获取肿瘤组织样本时,可采用外周血ctDNA作为补充标本评估EGFR基因突变状态,以明确最可能从吉非替尼治疗中受益的NSCLC患者[25]。CFDA在2015年2月已批准吉非替尼说明书进行更新,在推荐所有NSCLC患者的肿瘤组织都应进行EGFR基因突变检测基础上,补充了如果肿瘤标本不可评估,则可使用从血液(血浆)标本中获得的ctDNA进行评估,以尽最大可能明确最可能从吉非替尼治疗中受益的NSCLC患者。
全血中血浆和血清均能分离出ctDNA,但通过和相匹配的血清样本比较,血浆中ctDNA有更高的检出率[26]。血浆cfDNA通常片段较短,且在血液中浓度非常低[27,28]。抽血后延迟血浆分离会导致血细胞裂解,释放出基因组DNA(gDNA)至血浆中;大量增加的gDNA会稀释肿瘤来源的ctDNA,使得突变难以检出[29]。因此在标本的采集、运输及储存过程中,防止游离DNA的降解是首要考虑的因素;其次,也应防止血液中白细胞的裂解,避免因野生型DNA背景的增加导致ctDNA中的EGFR基因突变无法检测。
为了采集到最佳血浆标本用于后续提取游离DNA进行EGFR基因突变检测,建议使用如下两种方法之一采集10 ml全血并进一步分离血浆:(1)用含有游离DNA保护剂及防细胞裂解保护剂的专用常温采血管采集全血后轻摇混匀,常温(6~30 ℃)放置不超过5~7 d;以足够的离心力将全血充分离心两次,分离出不含细胞成分的血浆,放置于–70 ℃冻存直至DNA抽提,或直接进入DNA抽提步骤。(2)用常规EDTA抗凝管(严禁使用肝素抗凝管)采集全血后,两小时内以足够的离心力将全血充分低温离心两次[30,31],分离出不含细胞成分的血浆,放置于–70 ℃冻存直至DNA抽提,或直接进入DNA抽提步骤。
考虑到临床实践的方便性,建议尽量采用第一种方案采集10 ml全血。血浆分离过程中应注意避免吸入白细胞。
因ctDNA含量低,为提高EGFR基因突变检出率,在临床允许的情况下增加血浆用量,建议使用10 ml全血分离出的血浆(4~5 ml)。外周血游离DNA的提取有很多方法,建议采用国家药监部门批准的、大容量血浆游离DNA分离试剂盒。
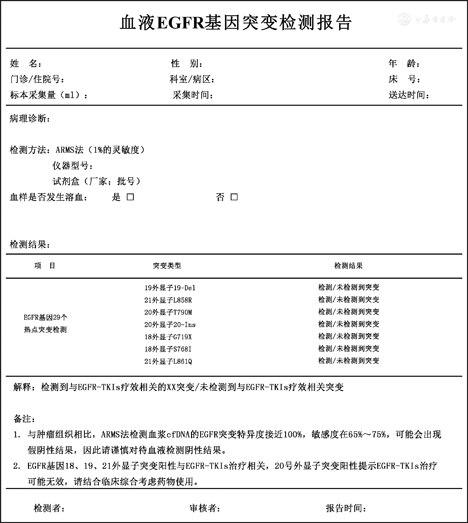
基因突变检测的报告内容应包括检测结果(建议包括明确的病理诊断及专业的书写)及对结果的诠释,且能够被肿瘤科医师或其他非病理专业的医师理解。其他内容应涵盖患者及标本基本信息、DNA质量、检测方法、检测试剂及仪器、相关临床意义以及在检测过程中出现的状况及不确定结果和因素,以供临床医师全面参考。检测报告模板见附件1。
所有专业化EGFR基因突变分子检测实验室,通过建立高效率的分子检测平台,优化流程,从接收标本到发出报告周期,建议不超过3~5个工作日,以及时满足临床的需求。
如前所述,血液检测EGFR基因突变用于筛选EGFR–TKIs适治患者是对组织检测的重要补充。血液检测还可以动态监测靶向治疗过程中肿瘤标志物变化,特别是可用于发现新出现的耐药相关基因变异[32]。
血液检测用于诊断晚期NSCLC有望实现,但诊断早期NSCLC有待检测技术不断的发展和进步。已有很多血液EGFR基因突变检测的方法的报道,如高分辨熔点曲线分析法(HRM)、变性高效液相色谱技术(DHPLC)、突变扩增阻滞系统(ARMS)法、二代测序、数字PCR方法等[17,32,33,34]。不同血浆游离核酸EGFR基因突变检测方法各有优势和劣势,但目前成熟且最常用的检测血液EGFR基因突变的方法是ARMS法,相较肿瘤组织EGFR基因突变检测,该方法检测血浆EGFR敏感型基因突变的敏感度在65%~75%,特异度在98%~100%[12,15,18]。数字PCR在血浆EGFR基因突变检测上具有较高的灵敏度和特异度[35,36],但目前报道的该方法检测EGFR基因突变位点相对有限(19外显子缺失,21号外显子L858R突变以及20号外显子T790M突变),仍处在摸索、累积经验阶段;其对操作人员和环境均有较高的要求,未来应用于临床尚需大规模临床数据验证。以二代测序技术(NGS)为代表的新技术拓展了基因突变检测的深度和广度[37,38],但其临床转化应用需要更多的数据积累。
未来,期待通过多元化基因检测平台及技术的不断发展与成熟,血液驱动基因突变检测可实现从定性到定量检测、从静态至动态检测、从单基因到多基因、基因组/外显子组检测的转变,从而推动NSCLC精准靶向治疗的不断进步。
当肿瘤组织难以获取时,血液是EGFR基因突变检测合适的替代生物材料,也是对可疑组织检测结果的补充;血液EGFR基因突变检测可用于对肺癌分子分型、疗效预测和疾病监测;目前成熟且最常用的血液EGFR基因突变检测方法是ARMS法;但是当肿瘤组织可以获取时,肿瘤组织仍应是优先选择的生物样本用于基因状态分析。随着检测技术的不断发展,期望在保证现有的检测特异性和预测准确性的基础上,不断提高血液EGFR基因突变检测的灵敏度,有助于整体上提高我国肺癌患者EGFR基因突变受检率,从而使更多的患者接受精准靶向治疗。NSCLC患者EGFR基因突变的检测流程见图1。
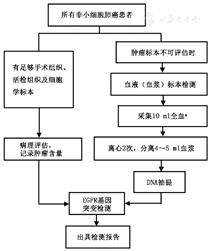

注:EDTA:乙二胺四乙酸;EGFR:表皮生长因子受体;a:专业采血管,常温放置不超过5~7 d或常规EDTA抗凝管2 h以内处理
本共识制订专家组成员(按姓名汉语拼音字母顺序排序):程颖(吉林省肿瘤医院肿瘤内科);韩宝惠(上海交通大学附属胸科医院肺内科);黄诚(福建省肿瘤医院胸部肿瘤科);梁智勇(北京协和医院病理科);林冬梅(北京大学肿瘤医院病理科);刘卫平(四川大学华西医院病理科);刘艳辉(广东省人民医院病理科);陆舜(上海交通大学附属胸科医院肿瘤内科);卢铀(四川大学华西医院胸部肿瘤科);邵建永(中山大学附属肿瘤医院病理科);宋勇(南京军区南京总医院呼吸内科);王洁(中国医学科学院肿瘤医院肿瘤内科);王哲海(山东肿瘤医院肿瘤内科);吴一龙(广东省人民医院胸部肿瘤科 广东省医学科学院 广东省肺癌研究所);应建明(中国医学科学院肿瘤医院病理科);张力(中山大学附属肿瘤医院肿瘤内科);张绪超(广东省人民医院、广东省医学科学院、广东省肺癌研究所);周彩存(同济大学附属上海市肺科医院肿瘤内科);周晓军(南京军区总医院病理科);周晓燕(复旦大学附属肿瘤医院病理科)