
版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
微生态学是指微生物生态学或微生境的生态学。人体微生态学特指研究人体微生物的生态学,可按人体部位分类,肠道微生态学较早且更多地受到关注[1]。人体微生态学的研究对象为微生物群(microbiota)或微生物组(microbiome),范畴包括微生物群(组)的特征及其与宿主的相互作用关系(图1)。本文旨在阐明微生物群(组)的概念,微生物群(组)与宿主的相互作用和调控方式,并对现有研究技术进行综述,指出人体微生态学研究领域亟需解决的问题和发展方向。
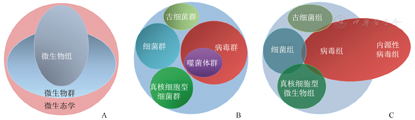

人体微生物群是对人体内部与体表所有微生物有机体的总称[2],其组成包括非细胞结构的病毒(包括噬菌体)、原核生物中的真细菌和古细菌,以及真核细胞微生物。与之对应,微生物群可以分为病毒群、细菌群、古细菌群和真核细胞型微生物群。而微生物组是特定时间特定生境(ecological niche)中微生物群所包含的基因序列(含同源序列)的总和[2,3,4]。两者不完全对应,微生物组的范围更广,特别是动物和植物微生物组。微生物组与其宿主基因组有重叠部分——主要是宿主基因组包含的与微生物同源的基因序列,特别是与病毒基因序列的同源部分。微生物组也可以相应地分为病毒组、细菌组、古细菌组、真核细胞型微生物组。相对于环境微生物组和动植物微生物组,目前研究人体微生物组所取得的成果最为丰硕。
人体微生物群的概念是从"人体正常菌群(normal flora)"演变而来的。一方面,人体无论是处在健康、亚健康还是疾病状态下,菌群都特定地存在于人体的一些部位,但多样性和丰度存在差异。因此,用中性词"微生物群"或"微生物组"取代"正常菌群"更合理。另一方面,以往对人体微生物的研究多关注细菌的作用,导致现阶段大多研究结果都是关于细菌组与宿主之间的关系。自美国病毒防御基金会的Norman G. Anderson等于2003年提出人类病毒组(human virome)以来,病毒组开始进入微生物组的研究范畴。由于病毒组的加入,人们对微生物组的定义范围已经不再局限于仅位于体表和某些腔道中,而是基于基因组序列的信息进行分析、挖掘。虽然目前国际和国内已有一部分关于病毒组的高水平研究,揭示病毒组与代谢性疾病、IBD、结直肠癌等疾病的关系[5,6,7,8],但相对于细菌组,对病毒组的认识才刚刚开始。
人体微生物群在黏膜器官分布最多,如口腔和肠道。据Ley等[3]和乔治亚大学Whitman等[9]估计,人结肠中所含的微生物约占人体全部微生物的70%。2016年初,魏兹曼研究所的Sender等[10,11]估算"标准体格"的成年男性结肠中微生物含量约为3.9×1013(人体自身的细胞数约为3×1013),而皮肤、口腔和女性阴道中仅1×1012左右。从种属水平上分析,肠道微生物群个体差异显著,但在门的水平上较为保守。人体肠道微生物群中丰度最高的是拟杆菌门和厚壁菌门,其次是变形菌门和放线菌门[12]。但其组成和丰度受宿主基因型、进化过程、饮食、地域和人为干预等因素的影响而会发生动态变化[13,14,15,16,17,18]。
通过微生物群与人体之间的基因交流而影响彼此的进化轨迹。2016年奥斯汀得克萨斯大学Moeller等[19]揭示了现代人和猿类并非简单地从环境中得到其肠道细菌,这些细菌在人科动物体内与宿主共同演化了数百万年之久并共同进化。
人体微生物群参与人体发育、生长和衰老的过程。如微生物群能够影响幼年时免疫系统的发育和建成[20];微生物群的一些代谢产物能影响大脑和神经系统的发育和功能,微生物群影响胃肠道的发育和血管系统的重构[21]。
微生物群与宿主对食品和药品代谢的调控深刻影响了人类的健康和疾病治疗,特别是对中药的药效影响巨大。肠道微生物群能通过初级和次级代谢途径产生大量的小分子物质(如短链脂肪酸等),这一生理过程依赖于宿主的饮食。虽然这些小分子物质中有很多被保留在肠道内,但还是有一些进入了循环系统并且被宿主进行化学修饰后发挥重要生理功能,最终通过尿液被排出体外[22]。
微生物组与宿主机体的相互调控主要涉及免疫系统、神经系统和内分泌系统。微生物组通过多种途径影响三大系统的功能,而机体生理状态的改变又影响微生物组的构成。此外,微生物组与宿主多种器官间还存在着重要的联系以进行相互调控作用,这种联系包括近年来备受关注的微生物组-肠-脑轴、微生物组-肠-肝轴和微生物组-肠-肺轴[23]。
大量研究表明微生物组与人体健康和疾病发生、发展存在密切关系,尤以肠道微生物组最为重要。人体微生物组与哮喘等超敏反应疾病,肥胖、糖尿病、肝脏疾病等代谢相关疾病,心脑血管疾病,IBS,IBD,慢性肾病,消化道肿瘤,自闭症、抑郁症和阿尔茨海默病等精神性疾病均有相关。历年文献报道与微生物组有关的人类疾病见表1。

人体微生物组相关疾病
人体微生物组相关疾病
| 部位 | 疾病 | 参考文献 | |
|---|---|---|---|
| 题名 | 刊发杂志信息 | ||
| 皮肤 | 痤疮 | Insights in the pathogenic potential of Propionibacterium acnes from its complete genome | Seminars in Cutaneous Medicine and Surgery, 2005,24(2): 67-72. |
| The complete genome sequence of Propionibacterium acnes, a commensal of human skin | Science, 2004, 305(5684): 671-673. | ||
| Acne is not associated with yet-uncultured bacteria | Journal of Clinical Microbiology, 2008, 46(10): 3355-3360. | ||
| 银屑病 | Substantial alterations of the cutaneous bacterial biota in psoriatic lesions | PLoS One, 2008, 3(7): e2719. | |
| Community differentiation of the cutaneous microbiota in psoriasis | Microbiome, 2013, 1(1): 31. | ||
| Structure and function of the human skin microbiome | Trends in Microbiology, 2013, 21(12): 660-668. | ||
| 过敏性皮炎 | Structure and function of the human skin microbiome | Trends in Microbiology, 2013, 21(12): 660-668. | |
| Skin microbiome: genomics-based insights into the diversity and role of skin microbes | Trends in Molecular Medicine, 2011, 17(6): 320-328. | ||
| Microbiome of affected and unaffected skin of patients with atopic dermatitis before and after emollient treatment | Journal of Drugs in Dermatology, 2014, 13(11):1365-1372. | ||
| 红斑痤疮 | Skin microbiome and skin disease: the example of rosacea | Journal of Clinical Gastroenterology, 2014, 48 Suppl 1:S85-86. | |
| 皮脂溢性皮炎 | Structure and function of the human skin microbiome | Trends in Microbiology, 2013, 21(12): 660-668. | |
| 口腔 | 龋齿 | Molecular analysis of bacterial species associated with childhood caries | Journal of Clinical Microbiology, 2002, 40(3):1001-1009. |
| Clonal analysis of the microbiota of severe early childhood caries | Caries Research, 2010, 44(5): 485-497. | ||
| 部位 | 疾病 | 参考文献 | |
|---|---|---|---|
| 题名 | 刊发杂志信息 | ||
| 龋齿 | Microbiota of severe early childhood caries before and after therapy | Journal of Dental Research, 2011, 90(11): 1298-1305. | |
| 齿龈炎 | Modulation of clinical expression of plaque-induced gingivitis: effects of personality traits, social support and stress | Journal of Clinical Periodontology, 2005, 32(11):1143-1150. | |
| 牙周感染 | Community as the unit of pathogenicity: an emerging concept as to the microbial pathogenesis of apical periodontitis | Oral Surgery, Oral Medicine, Oral Pathology, OralRadiology, and Endodontology, 2009, 107(6): 870-878. | |
| Root canal microbiota of teeth with chronic apical periodontitis | Journal of Clinical Microbiology, 2008, 46(11):3599-3606. | ||
| 牙周疾病 | Defining the healthy " core microbiome" of oral microbial communities | BMC Microbiology, 2009, 9(1): 259. | |
| Preliminary characterization of the oral microbiota of Chinese adults with and without gingivitis | BMC Oral Health, 2011, 11(1): 33. | ||
| Identification of candidate periodontal pathogens and beneficial species by quantitative 16S clonal analysis | Journal of Clinical Microbiology, 2005, 43(8):3944-3955. | ||
| 口臭 | Classification and examination of halitosis | International Dental Journal, 2002, 52(S5P1): 181-186. | |
| 口腔鳞状细胞癌 | The salivary microbiota as a diagnostic indicator of oral cancer: a descriptive, non-randomized study of cancer-free and oral squamous cell carcinoma subjects | Journal of Translational Medicine, 2005, 3(1): 27. | |
| Microbial diversity in saliva of oral squamous cell carcinoma | FEMS Immunology & Medical Microbiology, 2011,61(3): 269-277. | ||
| The microflora associated with human oral carcinomas | Oral Oncology, 1998, 34(4): 304-308. | ||
| 糖尿病 | The microbiological profiles of saliva, supragingival and subgingival plaque and dental caries in adults with and without type 2 diabetes mellitus | Oral Microbiology and Immunology, 2007, 22(3):175-181. | |
| Periodontal infection profiles in type 1 diabetes | Journal of Clinical Periodontology, 2006, 33(12): 855-862. | ||
| Diabetes and periodontal disease: a case-control study | Journal of Periodontology, 2005, 76(3): 418-425. | ||
| 动脉粥样硬化 | Detection of periodontal bacteria in atheromatous plaque by nested polymerase chain reaction | Journal of Periodontology, 2011, 82(10): 1469-1477. | |
| Cross-reactivity of GroEL antibodies with human heat shock protein 60 and quantification of pathogens in atherosclerosis | Oral Microbiology and Immunology, 2005, 20(5): 296-302. | ||
| Human oral, gut, and plaque microbiota in patients with atherosclerosis | Proceedings of the National Academy of Sciences,2010: 201011383. | ||
| 菌血症 | Incidence of bacteremia after chewing, tooth brushing and scaling in individuals with periodontal inflammation | Journal of Clinical Periodontology, 2006, 33(6): 401-407. | |
| 肥胖 | Is obesity an oral bacterial disease? | Journal of Dental Research, 2009, 88(6): 519-523. | |
| 胃肠道 | 过敏性疾病 | Gut microbiota, probiotics, and vitamin D: interrelated exposures influencing allergy, asthma, and obesity? | Journal of Allergy and Clinical Immunology, 2011,127(5): 1087-1094. |
| 类风湿性关节炎 | Coordination of tolerogenic immune responses by the commensal microbiota | Journal of Autoimmunity, 2010, 34(3): J220-J225. | |
| 炎症性肠病 | Low counts of Faecalibacterium prausnitzii in colitis microbiota | Inflammatory Bowel Diseases, 2009, 15(8): 1183-1189. | |
| Interactions between the host innate immune system and microbes in inflammatory bowel disease | Gastroenterology, 2011, 140(6): 1729-1737. | ||
| 乳糜泻 | Novel players in coeliac disease pathogenesis: role of the gut microbiota | Nature Reviews Gastroenterology & Hepatology, 2015,12(9): 497. | |
| 自闭症 | Microbiota modulate behavioral and physiological abnor-malities associated with neurodevelopmental disorders | Cell, 2013, 155(7): 1451-1463. | |
| 肝性脑病 | The relationship between intestinal microbiota and the central nervous system in normal gastrointestinal function and disease | Gastroenterology, 2009, 136(6): 2003-2014. | |
| 阿尔茨海默病 | Alterations of the human gut microbiome in liver cirrhosis | Nature, 2014, 513(7516): 59. | |
| Alzheimer′s disease and the microbiome | Frontiers in Cellular Neuroscience, 2013, 7: 153. | ||
| Mechanisms of molecular mimicry involving the microbiota in neurodegeneration | Journal of Alzheimer′s Disease, 2015, 45(2): 349-362. | ||
| 肥胖 | An obesity-associated gut microbiome with increased capacity for energy harvest | Nature, 2006, 444(7122): 1027. | |
| Metabolic endotoxemia initiates obesity and insulin resistance | Diabetes, 2007, 56(7): 1761-1772. | ||
| 非酒精性脂肪肝 | The role of the gut microbiota in nonalcoholic fatty liver disease | Nature Reviews Gastroenterology & Hepatology, 2010,7(12): 691. | |
| 糖尿病 | Metabolic endotoxemia initiates obesity and insulin resistance | Diabetes, 2007, 56(7): 1761-1772. | |
| 代谢综合征 | Metabolic syndrome and altered gut microbiota in mice lacking Toll-like receptor 5 | Science, 2010: 328(5975):228-231. | |
| 结直肠癌 | Structural segregation of gut microbiota between colorectal cancer patients and healthy volunteers | The ISME Journal, 2012, 6(2): 320. | |
| 部位 | 疾病 | 参考文献 | |
|---|---|---|---|
| 题名 | 刊发杂志信息 | ||
| 结直肠癌 | Gut microbiome development along the colorectal adenoma-carcinoma sequence | Nature Communications, 2015, 6: 6528. | |
| 脑卒中 | Commensal microbiota affects ischemic stroke outcome by regulating intestinal [gamma][delta] T cells | Nature Medicine, 2016, 22(5): 516-523. | |
| 艰难梭菌感染 | Species and genus level resolution analysis of gut microbiota in Clostridium difficile patients following fecal microbiota transplantation | Microbiome, 2014, 2(1): 13. | |
| Changes in predominant bacterial populations in human faeces with age and with Clostridium difficile infection | Journal of Medical Microbiology, 2002, 51(5): 448-454. | ||
| Shifting the balance: antibiotic effects on host-microbiota mutualism | Nature Reviews Microbiology, 2011, 9(4): 233. | ||
| 肠易激综合征 | A role for the gut microbiota in IBS | Nature Reviews Gastroenterology & Hepatology, 2014,11(8): 497. | |
| 冠状动脉疾病 | Gut microbiota and coronary artery disease | International Heart Journal, 2016, 57(6): 663-671. | |
| 肝硬化 | Gut microbiota and host metabolism in liver cirrhosis | World Journal of Gastroenterology, 2015, 21(41):11597. | |
| 泌尿道 | 尿石症 | The role of Oxalobacter formigenes colonization in calcium oxalate stone disease | Kidney International, 2013, 83(6): 1144-1149. |
| Enteric oxalate elimination is induced and oxalate is normalized in a mouse model of primary hyperoxaluria following intestinal colonization with Oxalobacter | American Journal of Physiology-Gastrointestinal andLiver Physiology, 2010, 300(3): G461-G469. | ||
| Rapid reversal of hyperoxaluria in a rat model after probiotic administration of Oxalobacter formigenes | The Journal of Urology, 2001, 166(4): 1487-1491. | ||
| 膀胱癌 | Evidence of uncultivated bacteria in the adult female bladder | Journal of Clinical Microbiology, 2012: JCM. 05852-11. | |
| Urine is not sterile: use of enhanced urine culture techniques to detect resident bacterial flora in the adult female bladder | Journal of Clinical Microbiology, 2013: JCM. 02876-13. | ||
| Human microbiota characterization in the course of renal transplantation | American Journal of Transplantation, 2014, 14(2):416-427. | ||
| 生殖道 | 细菌性阴道病 | Nonspecific vaginitis: diagnostic criteria and microbial and epidemiologic associations | The American Journal of Medicine, 1983, 74(1): 14-22. |
| Reliability of diagnosing bacterial vaginosis is improved by a standardized method of gram stain interpretation | Journal of Clinical Microbiology, 1991, 29(2): 297-301. | ||
| An international study of the interobserver variation between interpretations of vaginal smear criteria of bacterial vaginosis | Apmis, 2002, 110(11): 811-818. | ||
| Characteristics of three vaginal flora patterns assessed Gram stain among pregnant women | American Journal of Obstetrics and Gynecology, 1992,166(3): 938-944. | ||
| Suppressive antibacterial therapy with 0.75% metro-nidazole vaginal gel to prevent recurrent bacterial vaginosis | American Journal of Obstetrics and Gynecology, 2006,194(5): 1283-1289. | ||
| A randomized trial of the duration of therapy with metro-nidazole plus or minus azithromycin for treatment of symptomatic bacterial vaginosis | Clinical Infectious Diseases, 2007, 44(2): 213-219. | ||
| 良性前列腺增生 | Microbiota of male genital tract: impact on the health of man and his partner | Pharmacological Research, 2013, 69(1): 32-41. | |
| 艾滋病 | Vaginal lactobacilli, microbial flora, and risk of human immunodeficiency virus type 1 and sexually transmitted disease acquisition | Journal of Infectious Diseases, 1999, 180(6): 1863-1868. | |
| Bacterial vaginosis and disturbances of vaginal flora: association with increased acquisition of HIV | Aids, 1998, 12(13): 1699-1706. | ||
微生物群失调可能是由遗传倾向、感染、饮食和营养状况改变,以及使用抗生素等导致[21],改善肠道微生物组的方法主要有以下4种。
饮食能够显著影响肠道微生物群的组成,进而影响宿主的生理功能和免疫功能,以及对感染性疾病的敏感性[24]。
益生元(prebiotics),益生菌/益生生物(probiotics)和促生元(postbiotics)。近年的一些研究证明益生菌能够有效地调节肠道菌群,对于治疗腹泻、IBS、过敏性疾病、艰难梭菌感染等疾病具有一定的作用[25,26,27]。益生元能够刺激体内特定益生菌的增殖[28],而促生元为益生菌产生的能够正向调节生命活动的可溶性物质。但益生菌、益生元和促生元对特定疾病的作用尚需更多的随机对照试验证据。使用益生菌存在的安全性问题亦应重视[29],对于免疫功能缺陷或有肠漏症的人群不建议采用。
抗生素的使用不仅会导致细菌耐药,还会干扰正常菌群,导致机会致病菌的定植和致病,例如艰难梭菌感染。而噬菌体仅对个别菌株具有高度的特异性,能够感染并裂解这些特定的靶细菌,且对动植物无毒害作用,因此被认为是抗菌治疗的候选"药物"[30]。目前,已开展噬菌体疗法对H.pylori、艰难梭菌和具核梭菌等病原菌的感染等方面的研究。
通过粪菌移植将健康人粪便中的功能菌群移植到患者胃肠道内,重建新的肠道菌群,实现肠道和肠道外疾病的治疗。粪菌移植第一次在现代医学中应用是1958年由Eiseman等[31]用于治疗伪膜性肠炎。目前,国际和国内已有很多利用粪菌移植治疗感染性疾病或缓解疾病症状的成功案例,这些疾病包括IBD、IBS、代谢综合征等。
在人体微生态研究的起步阶段,相关研究集中于与疾病伴随的微生物组(主要是细菌组)多样性、种类和丰度的变化[32,33,34,35]。我国自20世纪中期开始与国际同步开展人体微生态学相关研究,代表学者主要有魏曦和康白。余 、谢少文等曾于20世纪60年代呼吁开展相关研究。经过几十年努力,已研发了22种微生态药品并在临床应用中取得了良好效果。近年来,我国微生物组研究也进入快速发展阶段,并取得了一系列重要进展。上海交通大学赵立平团队在肠道菌群与代谢功能或代谢相关疾病方面取得一些有价值的研究成果。该团队对一个四世同堂的中国家庭7位成员的粪便和尿液进行光谱学和微生物组分析,鉴定出肠道内参与人体代谢过程的一些重要细菌,并将20种尿液代谢产物与特定类型的细菌建立了关联[36]。在一项对2型糖尿病患者的研究中,该团队以宏基因组学技术结合代谢物分析,发现高膳食纤维饮食能够特定地促进产乙酸和丁酸的肠道共生菌株丰度的增高,增加代谢物中短链脂肪酸的含量,且相关菌株能够改善高脂饮食小鼠的体质量[37]。浙江大学团队发现肝硬化患者肠道菌群结构与患病状态相关,研究建立了世界上首个肝硬化患者肠道菌群基因集,阐明肝硬化患者肠道菌群的宏基因变化,并且建立了疾病预测模型,从肠道菌群紊乱的角度揭示了肝硬化的发展机制[38]。房静远团队经过多年的临床和实验室研究,发现具核梭杆菌在结直肠癌的发生中具有标志作用,且在结直肠癌化学疗法耐药中具有调控作用,并详细阐释了相应作用机制[39]。在微生态系统干预方面,建立了基于基因组的益生菌安全性评价体系[40]。
、谢少文等曾于20世纪60年代呼吁开展相关研究。经过几十年努力,已研发了22种微生态药品并在临床应用中取得了良好效果。近年来,我国微生物组研究也进入快速发展阶段,并取得了一系列重要进展。上海交通大学赵立平团队在肠道菌群与代谢功能或代谢相关疾病方面取得一些有价值的研究成果。该团队对一个四世同堂的中国家庭7位成员的粪便和尿液进行光谱学和微生物组分析,鉴定出肠道内参与人体代谢过程的一些重要细菌,并将20种尿液代谢产物与特定类型的细菌建立了关联[36]。在一项对2型糖尿病患者的研究中,该团队以宏基因组学技术结合代谢物分析,发现高膳食纤维饮食能够特定地促进产乙酸和丁酸的肠道共生菌株丰度的增高,增加代谢物中短链脂肪酸的含量,且相关菌株能够改善高脂饮食小鼠的体质量[37]。浙江大学团队发现肝硬化患者肠道菌群结构与患病状态相关,研究建立了世界上首个肝硬化患者肠道菌群基因集,阐明肝硬化患者肠道菌群的宏基因变化,并且建立了疾病预测模型,从肠道菌群紊乱的角度揭示了肝硬化的发展机制[38]。房静远团队经过多年的临床和实验室研究,发现具核梭杆菌在结直肠癌的发生中具有标志作用,且在结直肠癌化学疗法耐药中具有调控作用,并详细阐释了相应作用机制[39]。在微生态系统干预方面,建立了基于基因组的益生菌安全性评价体系[40]。
随着研究的逐步深入,国际上对微生物组的研究内涵、研究技术和应用方面也提出了新的要求。
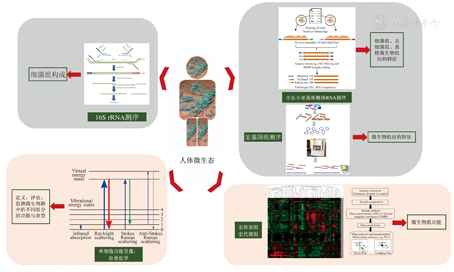
从精准诊断和医疗的角度来看,找出与疾病发生、发展有关的特定菌株,进而研究致病或缓解疾病的机制,对于人体微生态学而言无疑是一个值得深入研究的方向。目前常用的基于基因测序研究微生物群的方法只能间接展示微生物组特征,实际检测的灵敏度和准确度有待提高。这对微生物群中特定微生物的分离培养提出了要求。目前国际上已有一些研究团队对人体微生物群中的部分微生物进行分离培养并提出了培养组学(culturomics)概念[41]。基于培养的方法也将是微生物群(组)研究的"金标准"。但在培养方法尚未完善和规范之前,人体大多数微生物仍将难以实现大规模的分离和体外培养。在这一背景下,以核糖体RNA测序和宏基因组测序为代表的分析技术在肠道微生物群的研究中仍将发挥关键作用。
当前最为常用的16S rDNA扩增子测序,一般是对可变区V3、V3-V4或V6进行扩增并进行一代测序。基于这种测序方法,研究者能够利用较低的成本对微生物群中的细菌进行种水平以上的分类。但是测序的错误率较高且灵敏度低,另外,以DNA测序会忽略样品中生活状态的细菌和死亡细菌的差异。为了弥补这些缺憾,检测全长16S和18S rRNA测序的方法应运而生。利用核糖体小亚基(ribosomal small subunit,SSU)rRNA的多聚腺苷酸尾部进行反转录,合成对应于全长SSU rRNA的高质量、长读长cDNA用于构建测序文库。三代单分子测序能够很好地解决读长问题,但测序错误率高、成本高、通量低。利用二代测序的方法,获得全长SSU rRNA序列,能够将微生物群中的原核微生物和真核微生物鉴定到菌种水平,并且很大程度上降低测序错误率(降0.17%),提高测序通量且降低检测成本[42]。宏基因组测序的方法能够更大程度上提供微生物群中非细菌成分的信息,除了真核微生物之外,还有人体含量最高的微生物——病毒。该技术能够将人体的健康状态与微生物的生物化学途径进行关联,助力于进一步研究宿主-微生物群的相互作用关系[43]。宏基因组能够提供庞大的数据(测序成本较高),包括大量以往未能鉴定到的微生物,对序列的高效注释是宏基因组学发展的一个挑战。此外,如何对已鉴定的病毒组数据进行规范化分类和分析,也是宏基因组学发展过程中必须要解决的一大问题。
随着单细胞测序技术的兴起,应用于微生物群分析的单细胞测序也逐步在研究中被推广[44,45]。现有的宏基因组学、宏转录组学、宏代谢组学手段,DNA/RNA稳定的同位素探针,甚至培养组学方法,都不能直接揭示微生物群中不同组分之间的代谢互作关系。拉曼组学(Ramanome)——一种新型无标记的单细胞水平功能成像手段,能够检测微生物群体的功能和表型异质性,可以作为宏基因组学、宏转录组学和宏代谢组学的补充技术,来更好地诠释微生物群的状态[46]。在技术多元化和高速发展的前提下,对微生物群(组)数据进行规范和深度挖掘是有待解决的问题。
综上所述,基于人体微生态学的研究现状,在接下来的研究中亟需解决的问题主要有2个方面。①人体微生态学以研究细菌组为主转变为结合病毒组、真核微生物组等更为系统的微生物组研究;②在技术层面上,提倡采用规范化的宏基因组学结合宏转录组学、宏代谢组学,以及新型的单细胞成像技术,在揭示微生物群组成与丰度的基础上,进一步了解微生物群的转录、代谢状态、功能和表型异质性对人体健康或疾病的影响。见图2。

当涉及到利用动物模型研究人体微生态学中关于微生物群(组)与机体相互作用的机制时,如何科学地选择恰当的研究进化、发育或特定疾病的动物模型也是研究微生物群(组)所面临的问题。微生物群与宿主机体相互作用,特别是微生物群与人类共进化、共发育的机制是值得深入探索的一项科学问题。
在微生态学的应用层面,存在3个问题有待解决。①以微生物群为靶标的疾病诊断技术和产品,特别是人工智能与诊断标准优化相结合的精准医学诊断技术体系的建立;②以微生物群为靶标的干预技术和产品,包括天然健康微生物群的人工培养和人工合成微生物群扩增技术的研发,新型益生菌、益生元、促生元、噬菌体产品的研发;③各类微生物群相关诊断和干预技术,或产品有效性、安全性和质量控制的管理规范。
继2007年美国国立卫生研究院启动"人类微生物组计划"以来,国际上先后有多个国家和地区发起了相应的研究计划。经过10年的发展,在人类有关微生物组的研究取得突破性进展的同时,2016年5月13日,美国政府宣布启动"国家微生物组计划",这标志着微生态学研究由人体研究开始逐渐拓展到人类社会的各个层面,包括自然界的大气、水体和土壤。在我国,2007年至2014年的"973计划"和"863计划"中,投入7 000多万元用于支持肠道微生态的基础和技术研究。围绕"人体微生态"这一主题先后召开了香山科学会议"中国微生物组研究计划"学术研讨会(2015)和以"人体微生态与健康"为主题的国家自然科学基金委员会第144期双清论坛(2016)。至2017年12月,中国科学院重点部署"人体与环境健康的微生物组共性技术研究"暨"中国科学院微生物组计划"。这些研究计划必将推动人类对世界认识和改造的又一次跨越式发展。


























