
二代测序(next generation sequencing,NGS)的临床应用主要包括检测基因变异的全外显子组测序(whole exome sequencing,WES)和胎儿染色体非整倍体的无创产前筛查(non-invasive prenatal screening,NIPS)。本文回顾和概述了美国医学遗传与基因组学学会(American College of Medical Genetics and Genomics,ACMG)指导和规范NGS和NIPS临床应用的相关文件的发表和更新过程,这包括提出并更新专家共识性质的政策和立场声明,用于指导NGS技术的应用和变异解析,建立具有循证基础的实践资源以及标准指南来规范诊断和筛查,及时更新相应的文件和标准指南等。ACMG的这些文献对中国同行具有重要的参考价值,但由于中美在人群致病基因变异频率、社会经济状况和医疗实践方面的差异,直接采用这些文件和标准指南可能不切实际。希望本文有助于基于中国人群体的遗传病数据,结合国内对NGS技术的应用,探索建立符合中国国情和国际规范的NGS和NIPS的指导文件和标准指南,为遗传病患者及亲属提供优质、高效而安全的临床服务。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
2003年人类基因组测序的完成,促进了基因组技术向医学遗传学诊断和筛查的转化。二代测序(next generation sequencing,NGS)是通过DNA建库、特定序列捕获、序列扩增、大规模平行测序等步骤来实现高通量测序的技术。在临床将NGS应用于全外显子组测序(whole exome sequencing,WES)检测基因变异和无创产前筛查(non-invasive prenatal screening,NIPS)胎儿非整倍体的过程中,美国医学遗传和基因组学学会(American College of Medical Genetics and Genomics,ACMG)通过组织专家工作组针对关键问题进行系统的文献回顾,总结诊断和筛查方法的循证依据,提出了一系列的政策及立场声明(Policies and Position Statements)、专家共识(Expert Consensus)、实践资源(Practice Resources)以及实验室标准和指南(Standards and Guidelines)[1,2,3,4]。这些指导性文献促进了NGS技术临床应用的有序发展。而随着基因组技术应用的扩展和遗传病研究的快速进展,这些指导性文献得到了不断的更新,并逐步发展到建立各个遗传病种的临床实践指南(Clinical Practice Guidelines)。由于其他医学专科从业人员和公众缺乏对遗传病的了解,具有专业权威性的政策性文献不仅能够规范遗传病的诊治,而且可以推进遗传学知识在医学和社会教育中的普及。了解这些指导性文献的建立和更新过程及其在临床应用的效果,有助于针对中国人群体遗传病的特点建立适合于中国国情的诊断和筛查体系,为遗传病患者及其亲属提供优质、高效、安全的临床服务,并促进医学遗传学相关的科研合作。
人类基因组中约1%的蛋白质编码序列包含了大约85%与疾病相关的变异,最早应用外显子捕获技术结合NGS测序平台的WES在6例疑似为Bartter综合征的患者中检出了SLC26A3基因的致病变异。外显子捕获技术能够捕获95%的蛋白质编码序列,NGS的测序深度可达40×以上,基因变异检测的灵敏度和特异性均达到98%以上,这些结果证明WES有可能被用于临床基因检测[5]。2014年一项国际合作研究评估了基因组测序的数据分析、遗传病诊断、以及基因变异结果的解释和报告,结果发现测序分析的生物信息学技术逐渐趋同,但结果解释和报告尚有待进一步的发展和完善[6]。国内同行也验证了NGS在孟德尔遗传病诊断中的应用,并提出了规范基因检测报告的专家共识[7,8]。表1列出了ACMG自2013年起提出和更新的一系列针对NGS和WES的政策声明、实践资源、标准和指南[9,10,11,12,13,14,15,16,17,18,19,20,21,22]。
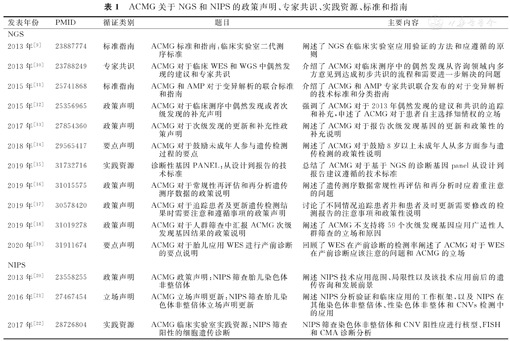
ACMG关于NGS和NIPS的政策声明、专家共识、实践资源、标准和指南
ACMG关于NGS和NIPS的政策声明、专家共识、实践资源、标准和指南
| 发表年份 | PMID | 循证类别 | 题目 | 主要内容 |
|---|---|---|---|---|
| NGS | ||||
| 2013年[9] | 23887774 | 标准指南 | ACMG标准和指南:临床实验室二代测序标准 | 阐述了NGS在临床实验室应用验证的方法和应遵循的原则 |
| 2013年[10] | 23788249 | 专家共识 | ACMG对于临床WES和WGS中偶然发现的建议和专家共识 | 介绍了ACMG对临床测序中的偶然发现从咨询领域内多方意见到达成初步共识的流程和需要进一步解决的问题 |
| 2015年[11] | 25741868 | 标准指南 | ACMG和AMP对于变异解析的联合标准和指南 | 介绍了ACMG和AMP专家共识联合发布的对于变异解析的技术标准和分类指南 |
| 2015年[12] | 25356965 | 政策声明 | ACMG对于临床测序中偶然发现或者次级发现的补充声明 | 强调了ACMG对于2013年偶然发现的建议和共识的追踪和补充,申述了ACMG对于患者自主选择知情权的立场 |
| 2017年[13] | 27854360 | 政策声明 | ACMG对于次级发现的更新和补充性政策声明 | 阐述了ACMG对于报告次级发现基因的更新和政策性的补充说明 |
| 2018年[14] | 29565417 | 要点声明 | ACMG对于鼓励未成年人参与遗传检测过程的要点 | 阐述了ACMG对于鼓励8岁以上未成年人从多方面参与遗传检测的政策性说明 |
| 2019年[15] | 31732716 | 实践资源 | 诊断性基因PANEL:从设计到报告的技术标准 | 总结了ACMG对于基于NGS的诊断基因panel从设计到报告建议遵循的技术标准 |
| 2019年[16] | 31015575 | 政策声明 | ACMG对于常规性再评估和再分析遗传测序数据的政策说明 | 阐述了遗传测序数据常规性再评估和再分析时应着重注意的问题 |
| 2019年[17] | 30578420 | 政策声明 | ACMG对于追踪患者及更新遗传检测结果时需要注意和遵循事项的政策声明 | 讨论了不同情况追踪患者并和患者及时更新需要修改的检测报告的注意事项和政策性说明 |
| 2019年[18] | 31019278 | 政策声明 | ACMG对于人群筛查中汇报ACMG次级发现基因结果的政策说明 | 阐述了ACMG不支持将59个次级发现基因应用广适性人群筛查的立场和原因 |
| 2020年[19] | 31911674 | 要点声明 | ACMG对于胎儿应用WES进行产前诊断的要点说明 | 回顾了WES在产前诊断的检测率阐述了ACMG对于WES在产前诊断应该注意的问题和ACMG的立场 |
| NIPS | ||||
| 2013年[20] | 23558255 | 政策声明 | ACMG政策声明:NIPS筛查胎儿染色体非整倍体 | 阐述NIPS技术应用范围、局限性以及该技术应用前后的遗传咨询和发展前景 |
| 2016年[21] | 27467454 | 立场声明 | ACMG立场声明更新:NIPS筛查胎儿染色体非整倍体立场声明更新 | 阐述NIPS分析验证和临床应用的工作框架,以及NIPS在其他染色体非整倍体、性染色体非整体和CNVs检测中的应用 |
| 2017年[22] | 28726804 | 实践资源 | ACMG临床实验室实践资源:NIPS筛查阳性的细胞遗传诊断 | NIPS筛查染色体非整倍体和CNV阳性应进行核型、FISH和CMA诊断分析 |
2013年ACMG提出的NGS实验室标准包括:(1)简介:简要描述了将NGS用于疾病相关基因包(disease-targeted gene panels)测序、WES和全基因组测序(whole genome sequencing,WGS)的3种方式;(2)概论:介绍了样品制备、DNA建库、分子条码、特定序列富集、测序平台和数据分析;(3)测序方法的前导考虑:主要涉及测序平台及其技术参数的选择;(4)方法的建立和验证:主要描述了测序平台的优化、数据分析以及临床有效性的验证;(5)分析标准:侧重于实验室质量控制、质量保证、人员资质、能力测试和数据保存等;(6)报告标准:主要涉及周转时间、数据解释、偶然发现变异的报告、报告格式和数据重新分析等[9]。
变异解析的技术标准和指南是基于ACMG和分子病理学会(Association of Molecular Pathology,AMP)的专家共识[11]。这一指南的主要目的是明确NGS检出变异的专属名词和详细分类。指南写作专家组对超过100个实验室进行了两轮问卷调查,征询变异的名词使用偏好和分类证据的要求、对11个实验室提供的变异分类方法进行了评估和综合、同时也通过文献检索了解了其他专业学会或工作组的序列变异分类指南。指南专家组建议用"变异(variant)"取代"突变(mutation)"和"多态性(polymorphism)",并将变异分为5类:致病性(pathogenic)、可能致病性(likely pathogenic)、意义不明(uncertain significance)、可能良性(likely benign)和良性(benign);将"可能致病"和"可能良性"的可能性定义为90%的概率以上。变异的命名采用人类基因组变异学会(Human Genome Variation Society)的标准命名法,同时也列表介绍了变异涉及的群体和病种数据库和分析工具。对变异分类的证据提出了详尽的标准,将致病性变异的证据分为非常强(pathogenic very strong,PVS)、强(pathogenic strong,PS)、中等(pathogenic moderate,PM)和支持(pathogenic supporting,PP)4个级别,且每个级别各有具体的细分(PVS1、PS1~4、PM1~6、PP1~4)。良性变异的证据可分为单一型(benign stand-alone)、强(benign strong)和支持(benign supporting)3个级别。专家组特别建立了综合这些证据划分5类变异的规则、证据框架和典型范例。变异分类还需要考虑遗传方式、族裔群体的发病率、携带者频率等因素,也应该包含结果、临床意义解释、方法、变异再分析、变异确证等。其他特殊的考虑则包括健康人群中偶然发现的变异、线粒体DNA变异、药理基因组变异、非孟德尔遗传的常见复杂疾病、体细胞变异等情况。一项研究对ACMG-AMP变异解析的技术标准和指南的效果进行了评估,结果表明接受测试的9个实验室变异解析的一致性为71%,说明仍需进一步改进以提高检测实验室分析报告的质量[23]。为进一步提高实验室检测报告的准确性和可靠性,2013年在美国人类基因组研究所的支持下,由包括29个国家的超过900名专家组成了Clinical Genome Resource(ClinGen)团队并针对现阶段存在于遗传病诊断过程中的问题成立了不同的工作组,其中的序列变异解读组致力于在遵循ACMG标准和指南的基础上进一步完善和规范对特定疾病在基因和变异水平的解读[24]。例如ClinGen于2018年提出了基于基因型和表型的相关性的综合评分来评价新发变异证据等级(PM2/PM6)的应用原则和适用范围(https://www.clinicalgenome.org/site/assets/files/3461/svi_proposal_for_de_novo_criteria_v1_0.pdf),其后ClinGen又于2020年提出了下调人群变异频率证据等级和同时增加由一个非常强(PVS)和一个支持性(PP)证据推导出的可能致病变异(likely pathogenic)的建议(https://clinicalgenome.org/site/assets/files/5182/pm2_-_svi_recommendation_-_approved_sept2020.pdf)。ACMG的标准和指南也列出了常用网站和公共数据库网址供分析者参考。
随着NGS临床应用的进展,ACMG继续提出了在儿科WES/WGS检测中鼓励大龄儿童和青年积极参与对话的声明[14]、针对诊断基因系列从设计到报告的技术标准[15]、针对测序结果进行再评估、再分析和再回访的声明[16,17]、以及WES在产前诊断中的要点说明等[15]。这些声明和标准的具体要求和要点都将会被纳入到临床检测的实践中。中国针对缺乏分子和临床诊断的先证者优先应用WES,致病变异的总体检出率达28.8%,是一项具有成本效益的临床实践[25]。
WES/WGS的应用改变了传统的基因检测模式,提高了诊断效率,但也有可能检出与送检临床指征无关、但对患者有医疗价值的偶然发现(incidental finding)或次级发现(secondary finding)的变异。ACMG组织了工作组评估并拟定了在临床检测中分析和报告偶然发现的原则,并列出了一份应该报告的偶然发现基因及其变异类别的清单。工作组在2012年3月ACMG年会的公开论坛上介绍了其原则和计划,并征求了反馈意见。2012年5月由ACMG理事会对其进行了初步审查,并随后根据多名专家和外部审稿人提出的反馈意见进行了修订。2013年ACMG理事会最终审核并发表了这份建议[10]。
该建议首先定义了诊断发现和偶然发现的概念,诊断发现被用于描述测序检出的与临床症状相关的致病基因变异;偶然发现则被用于描述在测序中检出的与临床指征不相关、但具有致病或可能致病性的基因变异。专家组还考虑了偶然发现的临床效用、局限、患者的选择偏好、对儿科患者的影响等。专家组的共识是提供一份"最低清单(a minimum list)",列出了对患者及其亲属有医疗益处的24种遗传病的56个相关基因。实验室在报告这些基因的偶然发现时应说明其与诊断发现的不同,并且根据现有文献和数据库解释偶然发现可能导致的临床症状。基于最低清单的结果对患者及其亲属的医疗益处,送检临床医师和实验室人员应熟悉临床测序的方法和局限性,有责任为患者提供全面的检测前和检测后咨询,提醒患者测序可能检出需要进一步评估的偶然发现,并且在不参考患者意愿和年龄的情况下报告最低清单的结果。鉴于基因组信息的复杂性,工作组专家和其他专业组织将至少每年完善和更新一次该清单建议的内容。
专家组也提出了如下的免责声明:本建议主要是作为临床遗传医师和其他医疗保健从业者的教育资源,促使其提供优质的医学遗传学服务。遵循这些建议并不一定能够确保成功的医疗结果。这些建议不应被视为涵盖了所有适当的医疗程序和测试,或排除了合理采用其他程序和测试获得相同的结果。在确定任何特定程序或测试的适当性时,遗传医师和其他临床医师应将自己的专业判断应用于个体患者所呈现的特定临床情况,并且在患者病历中记录下采用与建议不同程序和测试的理由。
对WES/WGS中必须报告偶然发现结果的建议被发表后,在医学遗传学及相关领域引发了争论和一系列的后续研究。反对方的观点认为这一强制性的要求不合理地增加了临床诊断实验室的工作量,对于所列病种的可治疗性和可预防性仍需要更多的证据,患者没有拒绝被告知的选择,以及对儿童告知成人后发病的病种是否违背现行的医学伦理等[26]。支持方的观点则强调了报告这些结果的医疗意义[27]。加拿大对遗传医师和遗传咨询师进行了问卷调查。95%的专业人士支持报告严重和可治疗的疾病的偶然发现,而对是否披露偶然发现的携带者、尤其是儿童携带者的问题缺乏共识。影响偶然发现报告最重要的考虑因素包括测试的准确性、致病证据的可靠性、预防及治疗的可行性、以及报告某些偶然发现(例如非亲子关系)的社会影响等[28]。另一项对遗传咨询师的问卷调查发现近半的咨询师会向患者报告和解释偶然发现的结果,大多数患者会接受偶然发现结果的报告,但也有患者希望拒绝接受这些信息[29]。一项回顾性研究调查了200个患者家庭对报告偶然发现的反应,该研究将WES中的偶然发现分为4类:隐性遗传病变异的携带者、易患晚发性遗传病、增加癌症风险的易感性和早发性疾病。结果发现93.5%的患者选择接受一个或多个类别的偶然发现,成年患者更可能选择接受偶然发现的结果[30]。AMP工作组则从临床实验室和患者的自主权的角度讨论了NGS技术的利弊、报告偶然发现的益处和危害,并将其与其他医学领域的实践进行比较,重申了遗传咨询保护患者自主权的重要性,强调有必要继续研究和讨论不同的政策对患者、临床和实验室从业者的影响[31]。更广泛的国际同行的批评和需要解决的问题包括适当的术语、充分的咨询或知情同意程序、选择退出的可能性、向未成年人的父母报告偶然发现的职业责任、患者自主权和治疗的可行性,这些问题存在内在关联并相互影响,需要在国际通行准则和具体案例之间取得平衡的解决方案,同时要纳入患者的观点来补充专业观点。以上研究说明需要更全面地综合了解报告偶然发现变异涉及的相关问题和各利益相关方的观点,最终提供一份有效的临床测序指南和相应的医疗保健政策[26,27,28,29,30,31,32]。
ACMG随后两度发表更新的政策声明[12,13],更新的建议包括:(1)临床WES/WGS应获得书面知情同意,并由具备资质的遗传专业人员说明测试的性质、结果解释的某些不确定性、隐私性、可能对其家庭成员的影响、以及可能检出与临床症状无直接关系的次级发现等。患者应了解无论具体临床症状如何,实验室将常规分析一组在医学上认为具有高度可干预性基因变异所致的疾病;(2)在知情同意的过程中应告知患者,如果需要,他们可以选择退出这类分析并了解这样做的后果;(3)根据医学伦理委员会的建议,ACMG理事会建议对儿童和成年人采用相同的政策,儿童的父母在咨询过程中可以选择退出这样的分析;(4)考虑到为患者咨询可干预的基因变异及其对应的遗传病的困难,也不可能让患者自主选择一组医学上可干预的基因,因此在常规分析中应该选择ACMG认可清单中的基因[12,13]。2016年ACMG工作组重审和更新了偶然或次级发现"最低清单"基因列表[13],新加了4个基因,去除了1个基因。目前需要报告次级发现的"最低清单"基因列表包括59个可干预的基因。未来的重点在于继续追踪临床实践中的结果和意见反馈,并研究报告次级发现的影响,以促进次级发现基因变异报告的标准化,同时对高外显性遗传病的风险通过既定的干预和预防措施降低发病率和死亡率[12,13]。最新的ACMG政策声明反对使用ACMG次级发现"最低清单"基因来进行普通人群的筛查[18]。
NIPS是应用二代测序技术分析孕妇外周血中游离DNA来筛查胎儿染色体非整倍体的方法。1997年Lo等[33]首次发现存在于孕妇外周血中的胎儿游离DNA可能被用于筛查胎儿的非整倍体异常。2008年,两个研究组分别使用NGS证明了NIPS检测非整倍体异常的可行性[34,35]。一项国际合作的病例-对照研究对NIPS的临床应用进行了验证,对4664名高危孕妇的测试发现NIPS对胎儿唐氏综合征(21三体)的检出率为98.6%,假阳性率为0.20%;对18-和13-三体的检出率分别为100%和91.7%,假阳性率分别为0.28%和0.97%[36]。这些结果表明NIPS能够有效地在产前筛查胎儿染色体非整倍体并且使侵入性产前诊断及相关风险减少95%[36,37,38,39]。这一技术不仅减轻了孕妇的焦虑,而且提高了医疗资源的利用率。
2013年,ACMG发表了NIPS筛查胎儿非整倍体的声明[20],强调NIPS的应用需要前瞻性临床试验结果以提供支持证据,指出NIPS技术平台对于全基因组、特定染色体或单核苷酸多态性的检测差异,并且对5个问题进行了重点阐述:(1)NIPS如何融入现有的产前筛查体系。临床数据表明,NIPS虽然具有高敏感性、高特异性和低假阳性等特点,但仍属于一种筛查方法。对胎儿进行确诊时,仍需要进行侵入性产前诊断;(2)NIPS局限于常见的三体如13-、18-、21-三体或性染色体数目异常,而无法筛查染色体结构异常、拷贝数变异和基因变异;(3)进行NIPS检测前后的遗传咨询应告知筛查的目的、技术特点和局限性、以及筛查高风险者后续的侵入性诊断方法;(4)筛查报告结果应清晰注明"筛查结果阳性"或"筛查结果低风险";(5)加强NIPS数据和生物信息学分析监管。NIPS实验室应该建立分析前、中、后质量控制标准和参与室间质评以保证结果的准确性。声明还介绍了NIPS相关的网站资源。2016年ACMG更新的立场声明认可新的证据支持NIPS取代传统方法筛选13-、18-和21-三体综合征,甚至可以延伸到性染色体非整倍性和选定的拷贝数变异[21],进一步印证了筛查方法的分析有效性、临床有效性和临床适用性。更新的声明对NIPS涉及的筛查前后的遗传咨询和后续检测的多面向过程、是否为所有孕妇提供筛查、如何避免和解释无结果的情况、是否将其用于筛查性染色体非整倍性和选定的拷贝数变异、以及多胎等特殊情况提出了具体的建议,同时也更新了NIPS相关的患者和检测方的网站资源。
2019年,ACMG发布了临床实验室对NIPS阳性结果进行细胞遗传学诊断的实践资源[22]。NIPS已被迅速纳入产前筛查的常规,所有筛查阳性结果的病例均建议通过产前诊断确认。细胞遗传学实验室通过绒毛膜或羊膜腔穿刺术获得胎儿样本进行染色体核型分析、荧光原位杂交和微阵列基因芯片分析明确诊断。NIPS阳性的患者在进行产前诊断时需了解以下信息:(1)胎儿游离DNA源自胎盘,有可能不代表胎儿;(2)筛查结果和诊断结果不一致的各种原因;(3)多条染色体异常或复合非整倍体可能与母体肿瘤有关。
NIPS由商业检测实验室主导,其应用范围已从筛查染色体非整倍性扩展到拷贝数变异和单基因变异[40,41,42]。技术的快速进步对相关标准和指南的建立构成了挑战。这些挑战包括需要更多的病例-对照系列的验证结果以提供支持证据和明确技术的局限性;需要积累临床数据以建立相应的参考资源;需要规范实验室的技术要求和报告标准;需要融入现行妇产科和遗传专科的筛查和诊断流程等,这些均对孕妇的遗传咨询、筛查结果和诊断方法的有效衔接以及结果的解释提出了更高的要求[43]。ACMG通过提供立场声明来指导NIPS的临床应用,其他的专业组织,如国际妇产科超声学会等,也提出了有关NIPS产前检查的共识声明[44,45]。后续标准和指南的建立需要利益相关方的参与、支持证据的积累、产检流程的更新、外部审查以及利益冲突的披露,以确保其实质性内容和质量有助于改善NIPS的临床服务[46]。
NGS技术在临床的应用已彻底改变了传统的基因检测,并将进一步广泛和深入地影响医学实践,其新的应用包括新生儿和儿科重症监护室的NGS快速检测报告,在美国、中国和其他地方正在探索和开发的NGS临床检测等大型项目等,这表明基因组数据将影响未来更加多样化并不断增长的患者群体的医疗决策[47]。对母血中胎儿有核红细胞或滋养细胞的基因分析有可能被用于无创产前诊断[48,49]。最新研究通过磁激活细胞分选母体血液中的滋养细胞进行测试的方法实现了单细胞低覆盖率的WGS,虽然需要更多的数据以确定其敏感性和特异性,但该方法具有成为无创产前诊断的潜力[49]。此外对血液中游离DNA的检测也可能被用于对正常个体和患者群体肿瘤标志物的监测[50,51]。
本文简要回顾了ACMG指导和规范NGS和NIPS临床应用的发展和相关文献的更新过程,主要包括提出具有专家共识性质的政策和立场声明及时指导技术应用和变异解析,建立具有循证基础的实践资源以及标准和指南来规范诊断和筛查,同时针对挑战,引导新的研究课题来更新。目前国内的遗传诊断虽然均已有意识参照ACMG的相关文献,但对ACMG的政策声明、实践资源、标准和指南理解得不够透彻,而且直接照搬可能脱离国内的实际。例如,次级发现的疾病和基因选列应该根据中国人群中基因致病变异的频率、社会经济状况和医疗政策的差异综合考虑。建议中华医学会医学遗传学分会和中国医师协会遗传医师分会根据中国人群体的遗传病数据,结合国内的NGS技术应用的现状,借鉴ACMG的经验,探索建立符合中国实际和国际规范的NGS和NIPS指导性文献,协同制定多学科临床实践指南,为遗传病患者及其亲属提供优质、高效、安全的临床服务。
感谢徐湘民教授和龚瑶琴教授在本文写作过程中给予的指导和帮助
所有作者均声明不存在利益冲突



























