
血型鉴定是对红细胞血型进行确认的一种方法,近一个多世纪以来,血细胞凝集反应成为血型鉴定的主要手段。随着分子诊断技术的发展,已诞生多种红细胞血型鉴定的基因检测分型技术,如限制性酶切片段长度多态性聚合酶链反应(PCR-RFLP)、序列特异性引物PCR (PCR-SSP)、多重PCR、适时定量PCR、PCR-酶联免疫吸附试验(ELISA)、DNA测序及基因芯片等。有关红细胞血型鉴定的新技术在检测通量、速度、便捷性及检测成本方面,均比血清学方法更具优势。笔者拟就红细胞血型鉴定技术的研究进展进行综述,旨在让临床医师对目前红细胞血型鉴定技术有一个系统性认识。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
自ABO血型发现以来,临床输血治疗的安全性已得到极大提高。输血治疗也成为挽救患者生命的重要手段[1]。目前在人体中已发现的红细胞血型系统有30多种,包含近400种血型抗原[2]。20世纪90年代前,红细胞血型鉴定主要依赖于血清学方法。近20多年来,随着分子生物学技术的发展,各种以聚合酶链反应(polymerase chain reaction,PCR),基因测序及生物芯片技术为基础的新型红细胞血型鉴定方法迅速发展。笔者拟就红细胞血型鉴定技术的血清学方法及各种分子诊断技术的基因分型技术进行综述,旨在让临床医师在临床输血治疗中对目前红细胞血型鉴定技术有一个系统性的认识。
红细胞血型鉴定的血清学方法主要包括玻片法、微柱凝胶法、试管法及抗人球蛋白试验法等,其基本原理均是利用抗原、抗体特异性反应鉴定红细胞表面抗原或血清中相应抗体。
玻片法属于直接血凝试验,是将血清与待检血液样品滴加在玻片上,通过观察红细胞凝集情况,对红细胞血型进行判定。研究认为,待检血液样品与检测试剂以1∶2体积进行匹配为最佳检测比例[3]。该方法鉴定红细胞血型的优点在于操作简便、快速,可以在2 min内完成检测,适合户外应用,且成本较低,目前应用广泛。但该方法灵敏度欠佳,可应用于红细胞血型初检,最终红细胞血型鉴定需要借助其他方法或进行复检。
试管法的血凝试验是在试管中进行,操作上不及玻片法简便,需要对血液样品进行离心,对于不易识别的凝集血液样品还需要进行显微观察。试管法采用盐水凝集鉴定红细胞血型时,易存在非特异性干扰,导致正、反定型不符,而采用聚凝胺试剂可提高抗体反应的特异性[4]。聚凝胺法对红细胞血型结果的鉴定,从凝集现象出现至消失,对振摇力度及抗体反应时间都要求准确掌握,操作难度相对较大。对于弱凝集血液样品,红细胞血型鉴定耗时相对较长。
微柱凝胶法通过观察待检血液样品能否通过凝胶颗粒构成的分子筛,判断血液样品凝集情况,进而鉴定红细胞血型。该方法与玻片法、试管法(含聚凝胺法)等其他传统红细胞血型鉴定的血清学方法相比,样品用量少、自动化程度高,且具有更高的灵敏度和特异度,尤其对弱凝集及存在自身免疫抗体的血液样品,具有更高的红细胞血型鉴定准确率[5]。操作上,该方法结果容易判断,易于保存,但检测耗时相对较长,不适宜应用于急诊对红细胞血型的鉴定。
抗人球蛋白试验是检测血液样品中意外抗体或补体的重要手段,包括检测红细胞表面意外抗体的直接抗人球蛋白试验,以及检测血清中意外抗体的间接抗人球蛋白试验。该方法对红细胞血型的鉴定在诊断自身免疫性溶血与新生儿溶血方面具有独特优势。目前,以微柱凝胶法进行的抗人蛋白试验,更可提高红细胞血型鉴定或意外抗体检测的灵敏度与特异度[6]。
分子生物学技术的发展,为红细胞血型鉴定的临床应用,特别是对红细胞血型抗原定型困难的患者或稀有血型献血者等特殊群体提供了便利。患者红细胞血型基因鉴定主要应用于:鉴别新生儿溶血风险,弱或无效(抗-Doa、-Dob、-Jsa及-V/VS)抗体患者,近期接受输血的患者,被抗体致敏的红细胞血型患者,区分异种抗体和自身抗体,弱表达抗原(Fyb表型抗原)不能刺激患者抗体产生时,以及鉴别分子异常血清学(特别是Rh变异体)等。红细胞血型鉴定中的献血者抗原分型主要应用于:筛选阴性抗原献血者,检测弱或无效(抗-Doa、-Dob、-Jsa及-V/VS)抗体,为增加阴性抗原库和寻找稀有血型献血者而进行大量筛查及检测弱抗原编码的基因等。
限制性酶切片段长度多态性PCR(PCR-restriction fragment length polymorphism,PCR-RFLP)应用于红细胞血型鉴定涉及相关血型基因的PCR扩增,然后进行相关血型基因片段长度的多态性分析。Wagner等[7]利用PCR-RFLP技术对RhD相关血型基因型进行分析研究,首先进行高保真PCR,正向引物为:CCTGTCCCCATGATTCAGTTACC,引物位点位于124 831~124 854碱基位;反向引物为:CCTTTTTTTGTTTGTTTTTGGCGGTGC,引物位点位于1 312~1 330碱基位,65 ℃退火,68 ℃延长10 min,然后利用PstⅠ内切酶进行酶切反应(37 ℃反应3 h),最后进行电泳分析。RhD–性纯合子扩增片段中有3个PstⅠ酶切位点,酶切反应后生成4个酶切片段(1 888,564,397及179 bp),RhD+纯合子有1个PstⅠ酶切位点缺失,故酶切反应后生成3个酶切片段(1 888,744及397 bp),RhD+/RhD–杂合子包含744 bp与564 bp片段[7]。PCR-RFLP技术对相关血型基因片段进行检测,由于限制性内切酶仅针对特异性核酸酶切位点,所以对红细胞血型鉴定具有高度特异性,缺点在于耗时较长,至少需要6 h,且受酶切位点的限制,不适用于所有基因多态性检测,同时内切酶的使用也增加了检测成本。
序列特异性引物PCR(PCR-sequence specific primer,PCR-SSP)技术根据红细胞血型等位基因中的单核苷酸多态性(single-nucleotide polymorphism,SNP),设计特异性引物,每对引物中的其中一条引物可以和每个等位基因结合,另一条引物仅能和等位基因中可以配对的等位序列结合,而其他等位基因则不能配对进行PCR扩增。Wagner与Flegel[7]采用该技术对RhD基因型进行分析研究,PCR正向引物为:GTTTGGCATCATAGGAGATTTGGC,引物位点位于120 101~120 124碱基位;反向引物为:CTCCCCCGATTTTAGCCAAGAA,引物位点位于6~27碱基位;60 ℃退火,68 ℃延长20 min,最后进行电泳分析。该技术通过引物序列的特异性设计,保证对红细胞血型等位基因检测的特异性,为提高技术的准确性,通常需要在反应体系中加入内参物,即混入另一对随机引物,该随机引物可以和任意血液样品的DNA配对结合进行扩增反应,从而以此判断PCR是否正常进行[8,9]。与PCR-RLFP技术相比,PCR-SSP技术对红细胞血型等位基因检测不需要进行内切酶反应,简化了操作程序,节约了检测成本,检测时间更多依赖于所扩增红细胞血型等位基因片段长度,但在判断纯合子与杂合子方面不及PCR-RFLP的特异度高。
PCR-RLFP与PCR-SSP技术均仅针对红细胞血型的单个等位基因为检测靶标,不能同时对多个基因进行检测。多重PCR(multiplex PCR)可在同一反应体系中针对红细胞血型的不同等位基因检测靶标设计多对引物,提高了检测通量[10]。Palacajornsuk等[11]利用多重PCR技术对来自10个血型系统的17个多态性位点进行分析,首先对血液样品进行多重PCR预扩增,将17个检测靶标分为3个反应体系,反应体系1检测Duffy和Dombrock血型系统,反应体系2检测Colton、Diego、Kidd、Landsteiner-Wiener、Lutheran、MN及Scianna血型系统,反应体系3检测Kell与Ss血型血统。红细胞血型鉴定的PCR反应时间需在2 h内完成,反应产物需要加入ExoSAP-IT PCR产物纯化试剂盒进一步纯化,以清除剩余脱氧核糖三磷酸核苷(deoxy-ribonucleoside triphosphate,dNTP)与引物。最后反应体系1和3体系以1.2%琼脂糖凝胶进行电泳分析,反应体系2因为多重产物大小差异小,以8%琼脂糖凝胶进行电泳分析,具有更高的分辨率。随后将预扩增产物进行多态性分析,探针引物为24个寡核苷酸链,每个引物3'末端为多态性位点的5'末端处,同时每个引物的5'端加有不同长度的聚腺苷酸(具有5个A碱基的长度差别),以避免PCR产物在最后分析时发生重叠。反应体系中加入4种荧光标记的双脱氧核糖三磷酸核苷(dideoxy-ibonucleoside triphosphate,ddNTP)[A=dR6G,绿;C=dTAMRA,黑;G=dR110,蓝;T(U)=dROX,红],PCR反应后加入虾碱性磷酸酶处理,最后使用测序仪进行SNP分析。Palacajornsuk等[11]利用多重PCR技术对29份血液样品进行检测,包括纯合子与杂合子的多个SNP分析均得到准确结果。
多重PCR技术虽然具有检测通量优势,但同时也存在显著缺点:①多对引物的退火、延伸及变性温度要求保持较为一致,否则PCR扩增反应不能正常进行,这一方面限制了检测通量,另一方面也增加了PCR检测的假阳性率或假阴性率;②多个PCR产物在进行电泳分析时,由于多个扩增产物的存在,且各种反应产物的量存在差别,给结果判断带来困难,对分析仪器的灵敏度也要求更高;③为了更好的分析产物片段长度,各血型基因设计的扩增片段长度需要有差别,所以在探针引物设计时,需要另加入不同长度的非特异寡核苷酸片段,以区别产物片段长度;④该反应需要反复进行PCR扩增反应及血液样品处理,操作程序复杂,耗时较长,红细胞血型鉴定一般需在6 h以上。因此,多重PCR技术虽然提高了红细胞血型等位基因检测通量,但仍受到多方面因素的限制,未能实现真正意义上的高通量检测。
适时定量PCR(real time PCR)技术主要特点在于可适时、动态检测红细胞血型等位基因靶标。Araújo等[12]采用2种荧光探针方法对100份血液样品进行检测,采用杂交探针方法检测了KEL1/KEL2、JK1/JK2和RHE/RHe(外显子5),采用SYBR Green I(一种结合于所有双链DNA双螺旋小沟区域的具有绿色激发波长的染料)探针方法检测了RHC外显子1,RHc外显子2,RHD内含子4、外显子7和4,扩增片段长度为96~600 bp,整个反应在1 h内完成[12]。该技术较PCR-RFLP及PCR-SSP技术在红细胞血型鉴定检测时间方面具有明显优势,程序更加简便,且能适时定量检测,但在检测通量方面不及多重PCR技术高。
PCR-酶联免疫吸附试验(enzyme linked immunosorbent assay,ELISA)技术的主要特征是结合了PCR与ELISA的反应原理,通过PCR对红细胞血型等位基因检测靶标进行扩增,随后将扩增产物用于后续反应检测。St-Louis等[13]采用该技术对RHD(β-Actin、外显子5和9),RHC/RHc(内含子2、外显子2、人类生长激素),RHE(外显子5和4),RHe(外显子5和4),RHK/RHk(外显子6、β-Actin),Fya/b(外显子1、人类生长激素)及JKa/b(外显子4、人类生长激素)进行检测。检测过程中通过PCR扩增引入地高辛标记的dUTP,随后在ELISA反应时首先在微孔板上包被链霉亲和素(streptavidin),再偶联上生物素标记的寡核苷酸探针(5'-biotinylated specific oligonucleotides)[13]。PCR产物加入微孔板,同时加入NaOH溶液,在碱性条件下PCR产物变性分解成单链DNA。孵育数分钟后加入中和缓冲液,使PCR产物与探针结合。随后加入酶标记的抗地高辛抗体,与PCR产物上的地高辛发生抗原、抗体结合反应,最后再以显色剂(邻苯二胺)显色,通过酶标仪进行检测[13]。该技术的最大优势是红细胞血型鉴定的灵敏度高,通过PCR与ELISA反应,2次提高灵敏度,特别适用于微量血液样品的红细胞血型检测。其不足之处也显而易见,操作复杂,耗时较长,且由于PCR与ELISA反应都存在假阳性率,所以该技术的特异度有一定限制,St-Louis等[13]研究中基因型与表型符合率为99.7%,基因型结果的假阳性率为3%,检测时间在6 h以上。
Sanger测序技术又被称为双脱氧碱基终止法,以特定位点作为DNA延伸反应的起点,在反应体系中加入不同荧光或其他方式标记的ddNTP,使延伸反应随机终止,随后对反应物进行电泳分析,以产物片段长度判断反应终止位点,通过检测标记物类型,判断终止碱基类别,通过高通量检测不同片段长度的末端碱基,进而达到测序的目的。1997年,Lin等[14]采用Sanger测序技术对人及其他8种灵长类动物的45份血液样品的A型转移酶互补DNA 56~742位的181 bp核苷酸片段进行分析发现,黑猩猩与绿猴的该片段包含2个MvaⅠ与1个HhaⅠ限制性酶切位点,人类该片段包含1个MvaⅠ与1个HhaⅠ限制性酶切位点,而其他灵长类动物包含1个MvaⅠ和2个HhaⅠ酶切位点位点,通过这2个酶切位点可以将人类与其他灵长类动物分开,该分析结果与PCR-RFLP技术的红细胞血型鉴定结果一致。梁延连等[15]通过Sanger测序技术对来自55份血液样品的MN血型GYPA基因的1~7外显子全长314~543 bp进行测序分析,其分析结果与血清学方法及PCR-SSP技术的分析结果完全一致。Sanger测序技术应用于红细胞血型鉴定准确性高,应用广泛,缺点在于检测速度不高,在检测通量方面不具有优势。
美国Illumina公司推出的Hiseq平台被称为第二代测序技术,即大规模平行测序技术((massively parallel signature sequencing,MPSS),其核心技术是将桥式PCR与单个核苷酸延伸技术相结合。首先将血液样品DNA通过超声打断成200~500 bp长度的小片段,并在这些小片段的两端加上接头,构建成单链DNA文库。其次是使单链DNA文库与微孔上的探针结合,然后进行桥式PCR扩增,最后进行测序反应。MPSS测序反应是以4种荧光标记的dNTP为原料进行反应,当1个dNTP被添加到延伸链上后,未反应的其他dNTP与聚合酶均被清洗掉,然后进行荧光信号检测。2013年,Cvejic等[16]采用血清学方法从35万人群中筛选出69份Vel阴性和20份Vel弱阳性血液样品,然后以Hiseq平台对Vel血型SMIM1基因进行测序分析的结果发现,69份Vel阴性血液样品中63个为纯合子基因,这些血液样品的SMIM1基因中有17个核苷酸序列存在缺失,在20份Vel弱阳性血液样品中,有19个为杂合子基因,而其余基因则是甲硫氨酸51突变引起的杂合子基因。MPSS技术对红细胞血型等位基因检测中的延伸反应和测序分析可同时进行,较Sanger测序技术大大提高了测序速度,可达600 Gb/run,测序通量上也具备优势,一次反应可同时检测16份血液样品。
焦磷酸测序原理不同于Sanger测序,为一系列酶促级联反应,反应中涉及多种酶的参与。PCR延伸反应中每个dNTP的聚合将释放一个焦磷酸,释放的焦磷酸在硫酸化酶的催化下与5'-磷酰硫酸腺苷(adenosine 5'-phosphosulfate,APS)反应生成ATP,产生的ATP又在荧光素酶(luciferase)的催化下与荧光素结合,生成氧化荧光素,同时产生可见光,进而可通过检测光强判断PCR的发生情况。PCR反应中每次加入1个核苷酸碱基,且4种碱基依顺序加入,这样反应与测序同时进行。Jaissle等[17]利用焦磷酸测序技术对来自8份血液样品3个血型系统进行分析,包括KEL血型KEL基因的1~19外显子,JR血型ABCG2基因的2~16外显子,以及LAN血型ABCB6基因的1~19外显子,每份血液样品含有53个外显子,设计了46个PCR扩增子,序列长度为366~576 bp,通过焦磷酸测序技术同时对8份血液样品的368个扩增子进行测序分析。随着纳米技术的进步,焦磷酸测序技术应用于红细胞血型鉴定,相对更加便捷且准确性高,Jaissle等[17]认为,454焦磷酸测序技术可同时对50~150份血液样品的KEL、JR及LAN血型系统进行分析,在检测通量上较Sanger测序具有显著优势。
Ion Torrent测序平台为新一代的测序平台,其原理与焦磷酸测序略有区别。Ion Torrent测序芯片为一个多孔反应体系,同时还是一个高灵敏离子传感器。各种dNTP依次流过微孔,当dNTP与模板互补配对反应后,将释放1个H+,从而引起溶液的pH值改变,高灵敏离子传感器检测到pH值改变后,立即将化学信号转变成电信号。Fichou等[18]利用该测序平台对15个血型系统的18个基因进行分析,包括ABO血型系统的ABO基因,MNS血型系统的GYPA、GYPB及GYPE基因,LU血型系统的BCAM基因,Kell血型系统的KEL基因,Duffy血型系统的ACKR1基因,Kidd血型系统的SLC14A1基因,Diego血型系统的SLC4A1基因,Yt血型系统的ACHE基因,Scianna血型系统的ERMAP基因,Dombrock血型系统的ART4基因,Colton血型系统的AQP1基因,LW血型系统的ICAM4基因,Cromer血型系统的CD55基因,Knops血型系统的CR1基因,以及Rh血型系统的RHD与RHCE基因。目前,Ion Torrent测序技术红细胞血型鉴定的血液样本均来自于血型基因文库和PCR扩增产物,尚未对人群血液样品进行检测,还无法评价该测序技术的实际临床应用价值,但该测序技术以高灵敏度离子传感器检测H+变化替代荧光扫描,红细胞血型检测速度显著提高。对于检测通量,该技术也有显著优势,如Fichou等[18]研究中同时对来自15个血型系统的18个基因包含417个扩增子进行测序,检测通量较其他测序技术有明显进步。
生物芯片技术在检测通量方面具有显著优势,Hashmi等[19]设计了磁珠核酸微阵列芯片,对400份血液样品的11个血型系统(Duffy、Dombrock、Colton、LW、Diego、Scianna、Kidd、Kell、LU、MNS及Hemoglobin S血型系统)进行分析,该磁株核酸微阵列芯片可同时检测96份血液样品,耗时约4 h。另有研究采用多重PCR技术与微阵列技术相结合[20,21],实现同时对多个血型系统的SNP位点进行检测,Le Goff等[20]利用96孔板多重PCR检测的微阵列技术对293份血液样品的血型系统进行检测,Kell、Kidd、Duffy及MNS血型系统的检测准确率分别为100%,98.9%,97.7%与97.4%。Bugert等[22]将设计的Taqman芯片用于血小板抗原检测,包括人类血小板抗原(human platelet antigen,HPA)-1,-2,-3,-5及-15。此外,应用生物系统(Applied Biosystems)推出的TaqMan探针芯片也在红细胞血型鉴定方面得到应用,该技术将定量PCR技术与微阵列技术相结合,通过TaqMan探针设计,实现高通量红细胞血型分型[22,23]。近年来发展起来的液相芯片(flexible multi-analyte profiling,xMAP)技术在红细胞血型检测通量上更具有优势,该技术的微球种类数已达到500种,理论上可以同时检测500种红细胞血型等位基因靶标。Karpasitou等[24]将xMAP技术应用于红细胞血型分型,同时对10多种红细胞血型系统进行鉴定的结果显示,xMAP技术在检测通量、便捷性、检测速度及成本方面具有显著优势,见表1。
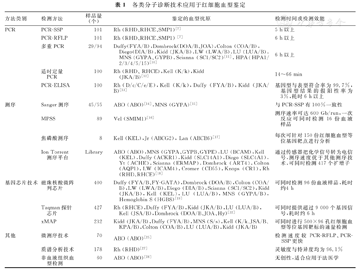
各类分子诊断技术应用于红细胞血型鉴定
各类分子诊断技术应用于红细胞血型鉴定
| 方法类别 | 检测方法 | 样品量(个) | 鉴定的血型抗原 | 检测时间或检测效能 |
|---|---|---|---|---|
| PCR | PCR-SSP | 101 | Rh (RHD、RHCE、SMP1)[7] | 5 h以上 |
| PCR-RFLP | 101 | Rh (RHD、RHCE、SMP1) [7] | 6 h以上 | |
| 多重PCR | 29/94 | Duffy(FYA/B),Dombrock(DOA/B、JOA),Colton (COA/B),Diego(DIA/B),Kidd (JKA/B),LW (LWA/B),LU (LUA/B),MNS (GYPA、GYPB),Scianna (SC1/SC2)[11],HPA(HPA1/2/3/4/5/15)[10] | 6 h以上 | |
| 适时定量 PCR | 100 | Rh (RHD、RHCE),Kell (K/k),Kidd (JKA/B)[12] | 14~66 min | |
| PCR-ELISA | 100 | Rh (D/c/C/e/E),Kell (K/k),Duffy (FYA/B),Kidd (JKA/B)[13] | 基因型与表型符合率为99.7%,基因型结果的假阳性率为3%,耗时6 h以上 | |
| 测序 | Sanger测序 | 45/55 | ABO (ABO)[14],MNS (GYPA)[15] | 与PCR-SSP有100%一致性 |
| MPSS | 89 | Vel (SMIM1)[16] | 测序速率可达600 Gb/run,一次反应可同时检测16份血液样品 | |
| 焦磷酸测序 | 8 | Kell (KEL),Jr (ABCG2), Lan (ABCB6)[17] | 每次可针对150份红细胞血型等位基因靶点进行分析 | |
| Ion Torrent测序平台 | Library | ABO (ABO),MNS (GYPA、GYPB、GYPE),LU (BCAM),Kell (KEL),Duffy (ACKR1),Kidd (SLC14A1),Diego (SLC4A1),Yt (ACHE),Scianna (ERMAP),Dombrock (ART4),Colton (AQP1),LW (ICAM4),Cromer (CD55),Knops (CR1),Rh (RHD、RHCE)[18] | 通过传感器把化学信号转为电信号,测序速度优于其他测序技术,可同时检测417个扩增子 | |
| 基因芯片技术 | 磁珠核酸微阵列芯片 | 400 | Duffy (FYA/B、FY-GATA),Dombrock (DOA/B),Colton (COA/ B),LW (LWA/B),Diego (DIA/B),Scianna (SC1/SC2),Kidd (JKA/B),Kell (KEL),LU (LUA/B),MNS (GYPA/B),Hemoglobin S (HGBS)[19] | 可同时检测96份血液样品,耗时约4 h |
| Taqman探针 芯片 | 427 | Rh (RHCE),Duffy (FYA/B),Kidd (JKA/B),LU (LUA/B),Kell (JSA/B),Dombrock (DOA/B、JOA、Hy)[22] | 可同时提供超过9 000个基因信号,耗时约6 h | |
| xMAP | 232 | Kidd (JKA/B),Duffy (FYA/B),MNS (S/s),Kell (K/k、JSA/B、KPA/B),Colton (COA/B),LU (LUA/B),Kidd (JKA/B) | 可同时进行500×96孔红细胞血型等位基因靶标的通量检测 | |
| 其他 | 微测序技术 | 70 | ABO (ABO)[25] | 检测速度较PCR-RFLP、PCR-SSP更快 |
| 质谱分析技术 | 178 | Rh (RHD)[27] | 灵敏度与特异度均为96.1% | |
| 非血液组织血型检测 | 60 | ABO (ABO)[28] | 无创性,适合应用于法医学 |
随着分子诊断技术的发展,以各种传统技术为基础的新型红细胞血型鉴定分子诊断技术也不断诞生,如以PCR为基础的微测序技术[25]和质谱分析技术[26,27]。另有研究者以骨组织为红细胞血型等位基因检测靶标,以吸附放散方法进行红细胞血型鉴定[28,29]。也有研究者将头发磨碎后,通过免疫组化方法进行红细胞血型鉴定,并具有较高的准确性[30]。上述非血液红细胞血型等位基因靶标的红细胞血型鉴定技术在临床应用较少,但在法医学中其意义更为显著。各类分子诊断技术应用于红细胞血型鉴定的情况,见表1。
红细胞血型鉴定的目的在于降低输血患者或胎儿溶血反应的概率,血型相容性将在更大程度上保证患者的输血安全。传统的血清学红细胞血型鉴定技术因为受到抗体制备的限制,且整体自动化程度不高,不可能达到同时对数十种血型系统的数百种抗原进行鉴定。随着分子诊断技术的发展,红细胞血型鉴定也正在掀起一场技术革命。采用现代分子诊断技术从基因分型的角度对红细胞血型进行鉴定,可达到同时对上百种抗原进行血型分析。以PCR、基因测序或生物芯片技术为基础的红细胞血型鉴定分子诊断技术的成本已有大幅度下降,且随着高通量技术的应用,红细胞血型的单个等位基因靶标检测成本相对更低。此外,红细胞血型鉴定的临床应用也受到相关法律、法规的限制,虽然目前大多数学者认为,血清学方法因其简单、方便及可靠的特点,仍适用于ABO与Rh血型鉴定,但本研究认为,随着红细胞血型鉴定技术的发展及相关法律、法规的调整,红细胞血型鉴定的分子生物学技术将在稀有血型库等建设方面得到实质性进展。






























