
红细胞血型的准确鉴定对临床输血安全至关重要。基因重组、碱基插入/缺失和单核苷酸取代等血型遗传学变异,均可导致红细胞血型基因多态性。血型遗传学变异的广度和复杂性,对红细胞血型鉴定提出了挑战。随着分子生物学技术的发展,血型基因分型、测序和基因芯片技术逐渐开始应用于红细胞血型分子诊断。笔者通过对红细胞血型基因多态性的分子基础、红细胞血型分子诊断技术的研究与应用进展进行综述,旨在阐明红细胞血型分子诊断技术对提高血型检测准确度和保障临床输血安全的重要意义。

版权归中华医学会所有。
未经授权,不得转载、摘编本刊文章,不得使用本刊的版式设计。
除非特别声明,本刊刊出的所有文章不代表中华医学会和本刊编委会的观点。
输血治疗是挽救患者生命的重要手段,对免疫血液学研究的深入,使临床输血治疗的安全性获得显著提高。尽管目前血清学检测仍是免疫血液学诊断的标准方法,但是由于多种血型抗原缺乏对应的抗血清,以及弱抗原可能导致假阴性结果,导致临床血型鉴定及精准输血困难。近年,血型分子诊断技术作为一种高度特异性血型表型预测的新方法迅速发展。红细胞血型基因多态性研究使得红细胞血型分子诊断技术的发展和血型基因新突变的鉴定成为可能,并建立了通过血型基因组DNA准确预测血型表型的方法。现今血型分子诊断技术的研究和应用进展迅速,为了加深基层和一线工作者对该技术的认识和关注,笔者拟就红细胞血型基因多态性的分子基础、红细胞血型分子诊断技术及其应用综述如下。
截至2019年8月,国际输血协会(International Society of Blood Transfusion,ISBT)认可的红细胞血型抗原总数为364个,其中326个红细胞血型抗原分属39个血型系统,第39号血型系统被列为红细胞血型的临时状态[1]。迄今几乎所有已知的红细胞血型抗原多态性的分子基础已被明确,并且开发出相应的分子检测技术。根据"千人基因组计划"获得的人类36个血型系统基因组变异信息,建立的Erythrogene数据库(www.erythrogene.com),共收录有43个红细胞血型抗原的210 412个等位基因多态性信息[2]。引起血型基因多态性的机制包括:基因重组、外显子复制、碱基插入/缺失、单核苷酸突变、调控区变异及DNA甲基化等[3]。
临床输血中,关注较多的红细胞血型系统包括:ABO、Rh、Kell、Duffy、Kidd和MNS等[4]。ABO血型系统中,A、B抗原是由9号染色体上ABO基因编码的α1,3-N-乙酰半乳糖胺转移酶和α1,3-D-半乳糖基转移酶分别催化前体物质H抗原转化形成,O抗原则是O等位基因在ABO基因第6外显子第261位发生单碱基G缺失,产生的一段仅由117个氨基酸组成的无活性蛋白。ABO基因长约20 kb,编码区前5个外显子共编码79个氨基酸;第6、7外显子编码275个氨基酸,其为决定糖基转移酶催化活性与特异性的主要部分。ABO基因多态性决定其血型抗原多样性,是产生弱A、弱B血型表型的根本原因,7种常见的ABO等位基因第6、7外显子的单核苷酸突变位点,见图1[5]。目前发现的ABO等位基因突变主要发生在编码区,但是某些亚型的分子机制仍未完全阐明。Cai等[6]发现,ABO基因的翻译起始点上游的启动子序列(位于-149和-2 bp之间)和5′端非翻译区中4个43 bp重复串联拷贝组成的微卫星序列,对ABO基因的转录活性至关重要。


Rh血型系统包含55个Rh抗原。人类Rh抗原由染色体1P34.3-36.4上2个紧密连锁且高度同源的等位基因RHD和RHCE编码,二者均有10个外显子,长度分别为57.4和58.2 kb。Rh抗原中,最具免疫原性的是D抗原。目前,已发现300多个RHD等位基因,其代表不同RhD血型变异型的分子机制,并且表现出抗原数量或质量改变的特征[7]。Zhang等[8]在132 479例无偿献血者中,筛检出血清学弱D表型45例(0.03%),并且在基因水平研究其分子机制,共检测出17个RHD突变型等位基因和2个新等位基因。Raud等[9]对RHD基因剪接位点变异的研究发现,11种由于基因剪接错误导致RHD基因转录本变异,包括RHD基因c.1065C>T沉默突变,其导致RHD基因第7外显子上形成新的剪接位点;RHD基因第9外显子c.1154G>A剪接位点突变,其可能导致RhD蛋白折叠错误,并且认为基因剪接突变可能是RhD变异型的一种常见分子机制。Kim等[10]对264例RhD血型阴性个体的研究发现,RHD基因全缺失、RHD-CE-D基因杂交、RHD基因点突变和RHD基因c.1227G>A突变,是引起受试者血清学检测结果RhD阴性的主要分子机制。
MNS血型系统包括49个血型抗原,其复杂程度仅次于Rh系统。位于人染色体4q28.2-31.1的GYPA、GYPB、GYPE基因调控MNS血型抗原的多态性。由单核苷酸突变所致的MNS血型抗原多态性已有10多种[11]。GYPA基因第1、2内含子上碱基突变也呈现出多态性特征。生物个体减数分裂过程中的碱基缺失、基因错位,以及高度同源的GYPA和GYPB等位基因之间的基因转换,导致EN(a-)、S-s-U-、Mk和Miltenberger等MNS血型抗原变异型的产生[12]。
Kell血型系统有36种血型抗原,其中K抗原的免疫原性最强。Kell抗原由人染色体7q33上的KEL基因编码。Ji等[13]对编码K抗原的KEL1基因第19外显子进行扩增、测序,并且将测序结果与编码k抗原的野生型KEL2基因进行序列对比发现,Kell血型抗原的多态性主要由单核苷酸突变引起。而KEL基因第10外显子的错义突变(c.1208G>A)导致Kell血型抗原减弱的Kmod亚型[14]。
Kidd血型系统由JK基因的2个共显性等位基因JK*01和JK*02编码,二者位于染色体18q12,3。Jk抗原由SLC14A1基因的2个共显性等位基因JKA和JKB编码,Jka/Jkb抗原的多态性与JK基因c.838A>G突变有关,并产生3种Jk血型表型:Jk(a+b-)、Jk(a-b+)和Jk(a+b+)[15]。
Duffy血型抗原由位于人染色体1q22-23的FY基因编码,其中Fya和Fyb抗原由FY*01和FY*02等位基因编码。由于FY基因启动子区域c.-46T>C纯合突变,破坏GATA-1红系转录因子结合位点,导致启动子活性减弱,从而产生Fy(a-b-)亚型[16]。
近几十年,红细胞血型分子诊断技术发展迅速,并且涌现出多种用于红细胞血型分子诊断的基因分型技术,如PCR-序列特异性引物(sequence specific primer,SSP),一、二代血型基因测序及微阵列(microarray)血型基因分型技术等。由于红细胞血型分子诊断新技术在检测通量、自动化效率方面较红细胞血型血清学检测方法更具优势,使其更广泛地作为血清学方法的替代或辅助手段。
PCR-SSP技术由于操作简单且高效,已成为目前实验室常用的红细胞血型分子诊断技术[17]。Yan等[18]采用PCR-SSP技术对中国汉族、塔吉克族、畲族、裕固族人群ABO、Rh、MNS、Duffy、Kidd、Yt、Scianna和Colton血型系统的20个血型抗原基因频率进行测定结果显示,不同人群的不同血型等位基因频率存在显著差异(P<0.05)。Lin等[19]利用PCR-SSP技术测定220例中国新疆回族个体的JK*A和JK*B等位基因频率分别为49.55%和50.45%,该结果接近汉族人群,但JK*A基因频率较中国藏族、维吾尔族及畲族显著降低(P<0.05)。随着高通量基因变异检测技术的应用和血型基因突变数据库的不断完善,使得利用遗传学信息鉴定红细胞血型抗原的方法在血站和输血科得以应用[20]。并且已开发出利用已知的血型基因单核苷酸多态性(single nucleotide polymorphism,SNP)进行准确基因分型的商品化试剂,检测数十种血型抗原[21]。然而,由于血型基因SNP检测是通过与已知突变位点的比对进行基因分型,而PCR-SSP技术仅对已知的特异性SNP位点有较好的检测,因此PCR-SSP技术对一些稀有等位基因或新突变很可能会漏检。另外,PCR-SSP技术对某些类型的DNA变异不敏感,特别是基因插入/缺失和结构变异。
DNA测序可以提供精确的核苷酸序列信息,是基因检测的金标准。一代血型基因测序技术,即Sanger测序技术,该技术将双脱氧核苷酸与荧光染料标记的碱基混合后,随机终止DNA链延伸,可以在读取基因序列后鉴定突变位点。目前,Sanger测序技术已经广泛应用于ABO血型亚型和其他稀有血型的鉴定,并且用于研究血型基因的新突变位点[22]。Sanger测序技术应用于红细胞血型分子诊断的准确率高、读取DNA序列长。然而,基因测序区域小,通量低和对基因转换等结构变异相对不敏感等不足,可导致在实践工作中使用Sanger测序技术进行全面的血型基因座测序存在一定局限[23]。
二代基因测序(next generation sequencing,NGS)技术在人类白细胞抗原(human leukocyte antigen,HLA)检测中的应用,有力地推动了其在红细胞血型分子诊断中的应用。NGS已应用于ABO和RHD基因的全长测序,可以实现在高通量DNA测序中,全面、精确的血型基因分子诊断。Sanger测序技术需要克隆突变基因后测序区分突变所在等位基因,NGS技术则是将突变基因通过桥式PCR技术使双链DNA片段,分别在固体支持基质上扩增形成单一DNA文库,使用特异性引物序列即可对突变的等位基因进行鉴定。Sanger测序技术中的读取基因序列长度受凝胶电泳泳道长度及凝胶质量等问题的限制,而NGS技术读取基因序列长度则由信噪比决定,通过提升荧光信号强度,提高信噪比可以获得更长的基因序列[24]。并且,NGS技术用于血型基因测序能够评估所有变异型等位基因。目前,通过NGS技术实现了人口规模的ABO血型分型。Lang等[25]分析欧洲169万人口ABO血型基因的研究结果,丰富了人类ABO等位基因频率的信息。Tounsi等[26]利用长距离-PCR技术扩增目的片段长且测序准确性高的优点,实现扩增完整的RHD基因序列,包括启动子、内含子和所有外显子。文献表明,通过NGS技术能够检测镰状细胞贫血(sickle cell anemia,SCA)患者RH等位基因是否变异,指导临床对稀有血型和Rh变异型患者制定输血策略,有效预防因输血导致的同种免疫发生[27]。因此,NGS技术可以补充传统血型血清学方法无法对血型抗原变异型进行有效鉴定的不足。
微阵列技术又称基因芯片技术,可以从基因组DNA中识别大量SNP。微阵列血型基因分型技术可通过检测1~2个SNP鉴定血型系统的大多数等位基因,并且可以灵活制定血型基因检测模块用于供血者血型鉴定。"芯片"测试基于与已通过多重PCR扩增的靶分子杂交,允许快速、并行、高效地检测数十种血型系统的多种等位基因SNP[28]。目前已有的商品化基因芯片主要包括HEA BeadChip、ID CORE XT和Bloodchip,前二者可对大多数常见的红细胞血型抗原进行基于阵列的检测,并且其检测效能已在来自欧洲的1 000份独立样本评估的大型试验中得到验证[29]。上述两种基因芯片都使用PCR扩增产物与芯片上携带的荧光基因探针杂交,通过荧光显微镜可检测多种红细胞抗原或变异体[30]。BloodChip可检测约213个血型等位基因,包括115个RHD基因型,以及血小板的24个等位基因[31]。3种基因芯片技术检测不同血型系统抗原的情况比较[29,30,31],见表1。
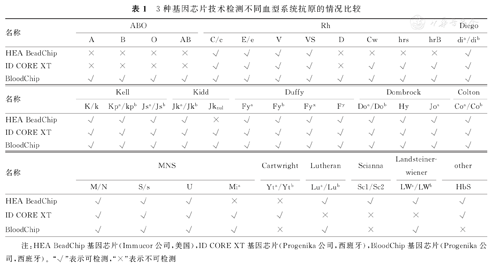
3种基因芯片技术检测不同血型系统抗原的情况比较
3种基因芯片技术检测不同血型系统抗原的情况比较
| 名称 | ABO | Rh | Diego | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | B | O | AB | C/c | E/e | V | VS | D | Cw | hrs | hrB | dia/dib | |
| HEA BeadChip | × | × | × | × | √ | √ | √ | √ | × | × | × | × | √ |
| ID CORE XT | × | × | × | × | √ | √ | √ | √ | × | √ | √ | √ | √ |
| BloodChip | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ |
| 名称 | Kell | Kidd | Duffy | Dombrock | Colton | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| K/k | Kpa/kpb | Jsa/Jsb | Jka/Jkb | Jknul | Fya | Fyb | Fyx | Fy | Doa/Dob | Hy | Joa | Coa/Cob | |
| HEA BeadChip | √ | √ | √ | √ | × | √ | √ | √ | √ | √ | √ | √ | √ |
| ID CORE XT | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ |
| BloodChip | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ | √ |
| 名称 | MNS | Cartwright | Lutheran | Scianna | Landsteiner- wiener | other | |||
|---|---|---|---|---|---|---|---|---|---|
| M/N | S/s | U | Mia | Yta/Ytb | Lua/Lub | Sc1/Sc2 | LWa/LWb | HbS | |
| HEA BeadChip | √ | √ | √ | × | × | √ | √ | √ | √ |
| ID CORE XT | √ | √ | √ | √ | √ | × | × | × | √ |
| BloodChip | √ | √ | √ | √ | × | √ | × | √ | × |
注:HEA BeadChip基因芯片(Immucor公司,美国),ID CORE XT基因芯片(Progenika公司,西班牙),BloodChip基因芯片(Progenika公司,西班牙)。"√"表示可检测,"×"表示不可检测
红细胞血型分子诊断技术的临床应用,主要解决血型精准鉴定和疑难血型的输血相容性问题[32]。红细胞血型分子诊断技术可用于筛查稀有血型和抗原阴性血型、疑难血型鉴定及产前新生儿溶血病诊断,以及为需要长期输血的贫血患者提供配合性输注策略。
Rios等[33]和Reid[34]发现对于存在Dombrock(Doa)、Colton(Coa)、Kell(Jsa、Kpa)及Diego(Dia)抗体的患者,利用血清学检测方法寻找对应抗原阴性供者非常困难。这是由于Doa、Coa、Jsa、Kpb、Dia等稀有血型抗原虽然有潜在临床意义,但是其抗原阴性献血者较为罕见,而且实验室常缺乏足量的抗血清试剂,因而利用血清学方法难以检出上述稀有血型抗体,并且这些抗体常合并存在其他同种抗体,使得对凝集试验结果的分析更加复杂。为了克服上述问题,红细胞血型分子诊断技术可以代替检出上述抗体血型血清学方法为患者快速筛选出抗原阴性的供者血液。目前,临床主要采用的高通量基因分型方法有SNaPshot测序[35]、TaqMan芯片技术[36]。此外,为了在血库中保存稀有血型供者血液,也需要利用红细胞血型分子诊断技术批量筛查献血者相应稀有血型基因而实现。
红细胞血型分子诊断技术是准确鉴定受自身抗体影响或血型血清学正反定型不符患者血型的有效工具[23]。此外,血型血清学检测为RhD阴性的育龄妇女或未成年女性在接受输血治疗时,血液中心需准确鉴定患者D抗原表型[37]。这是由于部分D变异型患者在接触D抗原免疫后可能产生D抗体,所以部分D变异型血型妇女在接受输血治疗时,应对其采用Rh免疫球蛋白治疗方案[37]。目前,传统的血型血清学方法不能准确区分弱D和部分D变异型,而通过测序RHD基因10个外显子及其邻近区域可以获得RHD基因编码区和非编码区序列多态性信息,达到准确鉴别部分D和弱D变异型的目的[38]。
胎儿血型鉴定对母婴血型不合引起的免疫性溶血性疾病的产前诊断有重要意义。由于羊膜穿刺术或绒毛膜绒毛取样鉴定胎儿血型时,存在妊娠终止和经胎盘出血增加母体抗体效价的风险。因此,上述侵入性操作,在临床应用具有一定局限性。近年来发现,在妊娠期以及产后3 d内,可以从母体外周血中提取游离的胎儿DNA,并采用高敏感度的实时PCR扩增等技术鉴定胎儿的血型基因[39]。根据这种产前无创胎儿血型鉴定技术的结果,可以对产妇实施有针对性地治疗措施,预防新生儿溶血病的发生。例如,对怀有RhD阳性胎儿的RhD阴性妇女进行免疫球蛋白治疗。这种产前无创胎儿血型鉴定技术,已成功地鉴定了胎儿ABO、RHD、RHCE、KEL和FY等血型基因,在血型基因水平上预测母婴血型不合与新生儿溶血病的发生风险[40]。
红细胞血型分子诊断技术可以在多方面指导临床配合性输血。首先对于有近期输血或多次输血史的患者,由于外周血中含有供者的血液细胞,使用经典的凝集试验无法准确鉴定患者血型,红细胞血型分子诊断技术可以克服该问题。对于此类患者,由于红细胞血型分子诊断技术检测的DNA目标是所有等位基因区域,即使存在少量献血者的DNA也会在PCR扩增过程中被患者的DNA竞争抑制,因而可获得准确的血型结果。其次,对于需要多次输血的SCA或β-地中海贫血患者,存在产生同种抗体的风险[41],建议在第1次输血前对其进行红细胞血型基因分型,以明确患者Rh、Duffy、Kidd、MNS等抗原表型,以便进行配合性输血减少输注无效的发生风险[42]。
随着对红细胞血型系统的深入研究,越来越多的红细胞血型基因分子基础被发现。同时,红细胞血型分子诊断技术存在应用基础和临床需求,相关技术包含简单易操作的基因分型技术和更为精准的测序技术等。血型分子诊断技术可应用于疑难血型鉴定、特殊血型抗原阴性血液的筛选、新儿血溶血病型的产前诊断及配合性输血,该技术的不断发展和应用,推动了输血医学的发展,并提高了输血安全。
所有作者均声明不存在利益冲突



























